
氏名:藤田 盛久
岐阜大学糖鎖生命コア研究所教授
2006年に筑波大学大学院生命環境科学研究科にて博士(理学)を取得。産業技術総合研究所、ジュネーブ大学、大阪大学にて博士研究員。2008年より大阪大学微生物病研究所にて特任助教、2012年より助教。2014年から江南大学生物工程学院糖化学生物技術教育部重点実験室教授。2022年より現職。糖鎖の生合成機構と改変に焦点を当て研究に取り組む。
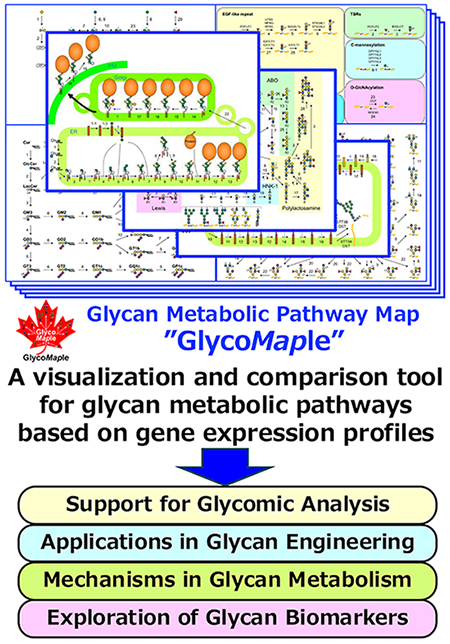
糖鎖は、修飾タンパク質の安定性、細胞内局在やターゲティング、細胞接着、細胞間相互作用、情報伝達など、多様な生命現象に関与する重要な分子である。鋳型をもたない糖鎖は、多彩な構造をとり、複数の糖転移酵素や糖加水分解酵素の連携により段階的に合成・分解される。糖鎖には異性体や電荷を有する構造が含まれることも多く、糖鎖構造の解析には高度な技術と経験が求められる。そのため、目的とする細胞や組織でどのような糖鎖が合成されているのかを把握することは容易ではない。こうした課題を解決する手段の一つとして、糖鎖生合成経路を簡便に可視化できるツール「GlycoMaple」が開発された。GlycoMapleは、細胞・組織のRNA-seqデータから得られる糖鎖関連遺伝子の発現情報に基づき、糖鎖の合成および分解経路を視覚的に表示し、生成されうる糖鎖構造を推定するものである。さらに、異なるサンプル間での比較によって、糖鎖代謝経路の違いを直感的に把握することも可能となる。本ツールは、糖鎖構造解析を補完する手段として有用であるだけでなく、糖鎖改変の設計や疾患マーカーの探索といった幅広い研究分野への応用が期待される。本稿では、GlycoMapleの開発背景、利用方法、活用事例、そして今後の展望について概説する。
糖鎖は、グルコース、ガラクトース、マンノース、フコース、シアル酸などの単糖が連なって構成される分子であり、遊離型として、あるいはタンパク質や脂質に結合した複合糖質として、細胞表面や細胞外分泌物中に広く存在する。ヒトを含む哺乳動物では、ヒアルロン酸のような遊離型糖鎖や、ガングリオシドに代表される糖脂質に加え、N型糖鎖、O型糖鎖、GPIアンカー、C-マンノースなどのタンパク質修飾型糖鎖を含む、多様な糖鎖が存在している1。これらの糖鎖は、修飾タンパク質の立体構造(フォールディング)や安定性、細胞内局在の制御に関与するほか、細胞接着、細胞間相互作用、シグナル伝達など、さまざまな生命現象に深く関わっている。一方で、糖鎖の変化は病的状態にも関連しており、ウイルス・細菌・毒素といった病原体の侵入経路として糖鎖が利用されたり、疾患に伴って細胞表面の糖鎖構造が変化することも知られている2,3。すなわち、糖鎖は健康と疾患の両側面において極めて重要な役割を果たしており、その構造の推定や決定は、基礎研究のみならず、医薬品開発や診断技術の分野においても不可欠である。
糖鎖は、核酸やタンパク質のような鋳型に基づく合成機構を持たないため、構造の予測や同定は容易ではない。糖鎖の合成は、小胞体およびゴルジ体に局在する200種類以上の糖転移酵素に加え、糖加水分解酵素、トランスポーター、基質合成酵素、さらには糖鎖結合タンパク質(レクチン)などを含む約950の糖鎖関連遺伝子産物により、段階的かつ競合的に制御されている4。さらに、これら酵素の基質特異性、発現量、細胞内局在、糖ヌクレオチドの供給、細胞の分化状態や疾患状態といった複数の因子が糖鎖構造に影響を与えるため、細胞がどのような糖鎖を持つのか、またそれがどのように形成されるのかを正確に予測するのは困難である。糖鎖構造の網羅的解析には、質量分析装置、高速液体クロマトグラフィー、レクチンアレイなどの技術が用いられているが、いずれも高度な技術と時間を要する。特に、分子量の大きな糖鎖や異性体、電荷を帯びた糖鎖の解析は困難であり、専門的な知識と経験が求められる。また、レクチンアレイは糖鎖の部分構造を比較するには有用であるものの、全体構造を明らかにするには限界がある。一方で、RNA-seqによって得られる糖鎖関連酵素遺伝子の発現プロファイルは、細胞や組織の状態を包括的に把握する手段として有用であるにもかかわらず、その情報を糖鎖構造の理解に直結させる方法は存在していなかった。
こうした背景のもと、糖鎖合成経路を可視化し、より簡便に糖鎖構造を推定することを目的として開発されたツールが「GlycoMaple」である5,6。GlycoMapleは、遺伝子発現データに基づき、それぞれの糖鎖合成・分解反応に関与する酵素遺伝子の発現量をパスウェイ図上に視覚的に表示し、細胞内で合成されうる糖鎖構造を推定するツールである(図 1)。これにより、目的の細胞や組織における糖鎖代謝経路の律速ステップや、サンプル間の経路の違いを簡便かつ俯瞰的に解析することが可能となる。
特定の細胞や組織でどのような糖鎖が合成されうるかを知ることは、細胞の状態や特性を知る上で重要な情報である。しかし、上述のように、質量分析装置や高速液体クロマトグラフィーを用いた糖鎖構造解析は技術的負担が大きく、一度に多種類の糖鎖構造を俯瞰的に把握することは困難である。一方、次世代シーケンシング技術の進展により、RNA-seqを用いた網羅的な遺伝子発現解析が広く普及した7。これにより、糖転移酵素、糖加水分解酵素、糖ヌクレオチドトランスポーターなど、糖鎖関連酵素遺伝子の発現量を高精度に取得することが可能となった。このことから、糖鎖の代謝経路(合成および分解)を図示し、各反応に関与する遺伝子の発現量を可視化することで、細胞や組織において合成されうる糖鎖構造を俯瞰的に予測できると考えた。
GlycoMapleの開発にあたっては、まずヒトにおける糖鎖生合成・分解に関与する約950の糖鎖関連遺伝子を整理した。対象とした遺伝子には、糖転移酵素、糖加水分解酵素、糖ヌクレオチド合成酵素、トランスポーター、転写調節因子、糖鎖認識分子(レクチン)などが含まれる。また、N型糖鎖、O型糖鎖、糖脂質、GPIアンカーなど、ヒトで既知の21種類の糖鎖代謝経路を網羅し、それぞれに対応した独自の経路マップを構築した。このマップは、symbol nomenclature for glycans(SNFG)に基づいて図示され、各反応は対応する酵素遺伝子によって規定されている(図 1)。各反応に関与する遺伝子の発現量(TPM値)を、矢印の色や太さによって視覚的に表現できるように設計した。初期設定では、1つの反応に複数の酵素が関与する場合、それらのうち最も発現量の高い遺伝子のTPM値を反映する。一方、複数のタンパク質が複合体として機能する反応については、構成因子のうち最も低い発現量を持つ遺伝子のTPM値を採用している。
本ツールの開発にあたっては、ヒト胎児性腎由来細胞(HEK293)をモデルとし、RNA-seqによる遺伝子発現データと、ムチン型O型糖鎖構造の実測データを比較・解析した5。その結果、TPM = 1を閾値とした場合、遺伝子発現から予測される糖鎖と、実際に検出された糖鎖構造とが良好に一致することが示された。このようにしてGlycoMapleは、遺伝子発現データに基づいて糖鎖代謝経路を直感的に把握し、理論的に合成されうる糖鎖構造を可視化するツールとして設計された。
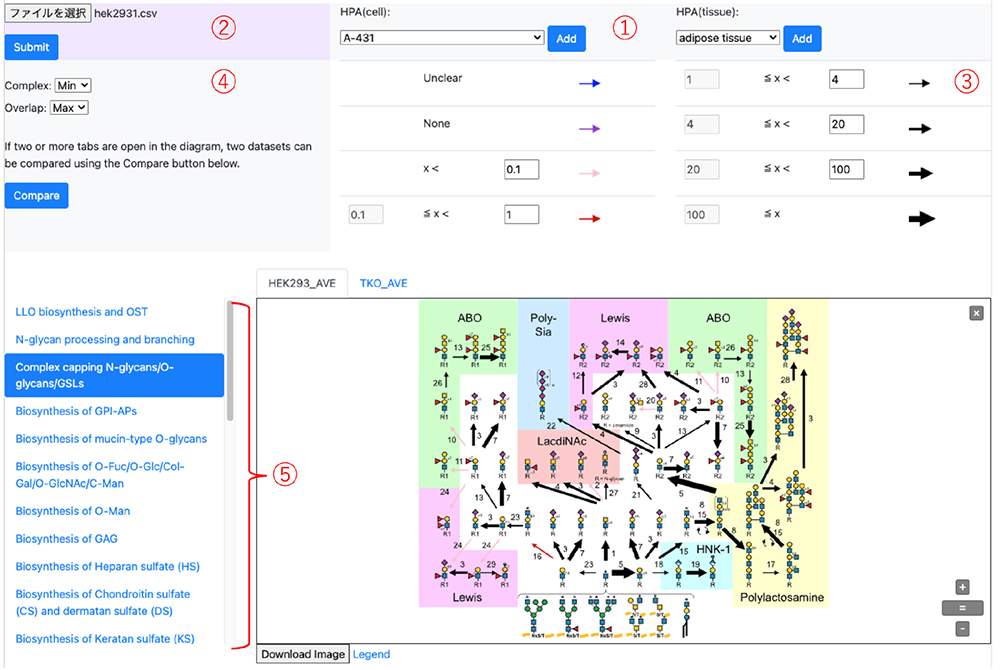
現在、GlycoMapleはGlyCosmosポータル(https://glycosmos.org/glycomaple)上で公開されており、Webブラウザ上で操作可能である8。ヒトGlycoMapleには、あらかじめ、The Human Protein Atlas(HPA)に収録されたヒト培養細胞や組織の遺伝子発現データが収録されており、目的の細胞あるいは組織を選択して、糖鎖代謝経路を可視化解析することができる(図 2-①)。また、自前で準備したRNA-seq解析結果をCSVファイルでアップロードして、解析を行うことも可能である(図 2-②)。詳しい利用方法は別文献を参照されたい9,10。
それぞれの反応を示す矢印の色や太さは、反応を担う酵素遺伝子の発現量(TPM)で規定されている(図 2-③)。初期設定では、上述のように、TPM = 1を閾値としており、それ以上の発現量を示す場合、黒色の矢印で示され、発現量に応じて矢印の太さが変化する。一方で、反応を担う酵素遺伝子の発現がTPM < 1の場合、細胞あるいは組織では発現が見られないと定義され、赤色(TPM < 1)あるいはピンク色(TPM < 0.1)で示される。これら閾値は、ユーザーの目的によって変更可能である。また、上述したように初期設定では、一つの反応に、複数の酵素が重複して関与している場合は、それらの酵素遺伝子の発現量の最大値、複数のタンパク質が複合体を形成している場合は、それらのタンパク質をコードする遺伝子の発現量の最小値が反映されるようになっている(図 2-④)。これらの設定も、ユーザーの目的によって、最小値、平均値、最大値、合計値への変更が可能である。
解析画面では、21種類の糖鎖代謝経路マップが選択可能であり、遺伝子発現データをもとに、各反応の矢印が動的に更新される(図 2-⑤)。矢印をクリックすると対応する遺伝子のHuman Genome Organization (HUGO) Gene Nomenclature Committee (HGNC) ページへのリンクが表示され、遺伝子の詳細情報にもアクセスできる。また、それぞれの経路に関わる遺伝子の発現は、経路の下にbar plotとして表示される5。さらに、2つ以上のサンプルを登録した場合には「Compare」機能が利用でき、任意の2サンプル間の糖鎖代謝経路を比較することが可能である。発現の増減に応じて矢印の色が変化し(緑=減少、ピンク=増加)、視覚的に比較解析を行うことができる。
GlycoMapleは遺伝子発現情報に基づいて糖鎖の合成・分解経路を俯瞰的に可視化できることから、糖鎖研究のさまざまな分野での活用が期待される(図 1)。以下に主な応用例を紹介する。
糖鎖の構造解析には質量分析が広く用いられているが、糖組成の同定は可能であるものの、糖鎖の結合様式までを正確に識別することは困難である。例えば、末端にシアル酸を含む糖鎖が存在する場合、α2,3結合かα2,6結合かを質量分析のデータだけで判別することは難しい。GlycoMapleでは、対象となる細胞や組織における糖鎖合成酵素の発現プロファイルを可視化することができるため、理論的に合成可能な糖鎖構造の候補をあらかじめ提示することができる。上述の例では、シアル酸転移酵素の発現パターンを確認することで、どちらの結合様式が優勢かを推定することが可能である。これにより、質量分析で得られたフラグメントと照合すべき構造の絞り込みが可能となり、糖鎖構造解析を理論的に補完する補助ツールとして機能する。
GlycoMapleは、細胞内の糖鎖代謝経路や構造を推定することができ、律速となる反応を特定することで、糖鎖改変の戦略設計にも活用できる。特定の糖鎖構造を増強あるいは抑制する目的で遺伝子改変を行う際、その効果を視覚的に評価することができる。例えば、糖転移酵素の過剰発現やノックアウトによって糖鎖経路がどのように変化するかを可視化し、効率的に目的の糖鎖修飾を達成するための設計指針を得ることができる。現在、HEK293細胞を用いた糖鎖関連遺伝子のノックアウト細胞ライブラリーが整備されつつあり5,11,12、GlycoMapleとの組み合わせによって各遺伝子改変の効果を体系的に評価することが可能となってきている。
糖鎖生合成の調節機構の解明においても、GlycoMapleは有用である。時系列あるいは条件変化に応じた酵素遺伝子の発現変動を視覚的に解析することで、律速酵素や代謝物供給の制御点を同定する手がかりが得られる5,13。例えば、N型糖鎖のマンノシダーゼ遺伝子(MAN1A1, MAN1A2, MAN1B1)をノックアウトした細胞では、ヒアルロン酸合成酵素HAS2の発現が著しく上昇し、ヒアルロン酸の産生が増加することが確認された5。また、LacNAcを含む糖脂質の増加も観察され、糖鎖合成経路間に代償的な調節やフィードバック機構が存在することが示唆された。
また、抗酸化ストレス処理を行った条件で遺伝子発現情報を取得し、GlycoMapleを用いて解析が行われている14。その結果、α1,6-フコース転移酵素FUT8の発現が著しく上昇しており、これに伴う糖鎖構造の変化が予測された。この予測は、レクチン染色によって実験的に検証され、GlycoMapleの有用性が裏付けられた。こうした例から、GlycoMapleは環境刺激や細胞状態の変化に応じた糖鎖修飾の動的制御を可視化し、仮説の立案や検証に役立つ解析ツールとして機能する。
GlycoMapleは、疾患に関連する糖鎖マーカーや治療標的の探索にも応用可能である。疾患組織と正常組織の遺伝子発現データを比較することで、疾患特異的な糖鎖合成経路の変化を可視化し、特異的な糖鎖構造の出現や酵素発現の異常を明らかにすることができる13,15。例えば、大腸がん組織と正常組織における糖鎖経路の比較では、MGAT5、B3GNT2、FUT3、ST6GAL1などの糖転移酵素遺伝子の発現変化が検出され、疾患特異的な糖鎖構造の形成に関与している可能性が示唆された5。グライコプロテオーム解析とGlycoMapleを組み合わせた統合的なアプローチは、疾患における糖鎖の機能解明、新たなバイオマーカーの同定、治療標的の探索に有用であると期待される。
GlycoMapleは、遺伝子発現データに基づいて糖鎖代謝経路を推定するツールであり、実際に存在する糖鎖構造を直接証明するものではない。mRNAの発現量と酵素活性との間には必ずしも直接的な相関があるとは限らず、タンパク質の細胞内局在、糖ヌクレオチドの供給、基質の存在量、細胞内輸送の状態など、複数の因子が糖鎖合成に影響を及ぼす。そのため、推定結果には偽陽性または偽陰性が含まれる可能性があり、得られた経路情報はあくまで仮説生成のための参考情報と位置づけるべきである5。実際の糖鎖構造を確認するには、質量分析などの実験的手法による検証が不可欠である。
現時点では、GlycoMapleは細胞や組織で合成されうる糖鎖構造を示すことができるが、実際にどの糖鎖がどの程度合成されているかを定量的に予測することは困難である。また、代謝経路におけるフィードバック制御などの動的挙動を反映することも難しいという制約がある。こうした課題を克服するために、近年では酵素学的パラメータや機械学習を活用し、糖鎖生合成プロセスを動的にシミュレーションする手法の開発が進められている16-18。特に、糖転移酵素の酵素活性や細胞内局在に関する情報を統合することで、より現実的な糖鎖合成過程を予測するモデルの構築が試みられている。我々のグループでも、まずN型糖鎖の分岐構造に関与する主要酵素群に着目し、酵素活性情報、遺伝子ノックアウト細胞における糖鎖構造データ、遺伝子発現データを統合することにより、糖鎖代謝経路を反映した動的予測モデルの構築に取り組んでいる6,19。また、マウス版のGlycoMaple(現在、限定公開中(論文投稿準備中))やシングルセルRNA-seqデータをGlycoMapleで解析したいというニーズも寄せられており、今後、利便性拡大を視野に入れて開発を進めていく必要がある。
GlycoMapleは、これまで解析が難しかった糖鎖を、遺伝子発現情報に基づいて簡便かつ俯瞰的に解析可能とした新しい可視化ツールである。複雑かつ専門性の高い糖鎖合成経路を直感的に理解できるよう設計されており、遺伝子発現データをアップロードするだけで、糖鎖代謝経路の全体像を把握することができる。このような特性から、異分野の研究者が糖鎖研究に参入するための足がかりとして利用していただけると幸いである。多くの生命現象や疾患に糖鎖が関係すると考えられ、GlycoMapleを利用することで、癌、免疫、感染症、幹細胞、創薬研究などの分野に広がることを期待したい。