
氏名:矢木 宏和
名古屋市立大学大学院薬学研究科 准教授/ 自然科学研究機構 生命創成探究センター 客員准教授
2003年名古屋市立大学薬学部卒業。2008年名古屋市立大学大学院薬学研究科・博士後期課程修了。その後、同研究科および自然科学研究機構生理学研究所において日本学術振興会特別研究員。2009年名古屋市立大学大学院薬学研究科、助教に着任し、講師、准教授を経て、2023年より、独立准教授として研究室を運営している。また2022年より、自然科学研究機構 生命創成探究センター 客員准教授を兼職。様々な生命現象を担う糖鎖の生合成過程および機能解明を目指して研究に従事している。

氏名:近藤 幸子
自然科学研究機構 生命創成探究センター 特任専門員
2005年 愛知教育大学大学院教育学研究科理科教育修士取得。2005年糖鎖構造解析技術を基盤とする名古屋市立大学発ベンチャー「グライエンス」に入社。2023年より現職。HPLCマップを用いた糖鎖構造解析に従事している。

氏名:加藤 晃一
自然科学研究機構 生命創成探究センター(ExCELLS)教授
1986年、東京大学薬学部卒業。1991年、同大学院博士課程修了(薬学博士)。同年より東京大学大学院薬学系研究科にて助手、講師を務め、2000年より名古屋市立大学大学院薬学研究科教授。2008年、自然科学研究機構 岡崎統合バイオサイエンスセンター教授に就任(後にExCELLSへ改組)、2018年より現職。現在、名古屋市立大学教授とExCELLS教授を兼務し、糖鎖修飾の機構を分子・細胞レベルで統合的に解明する研究を展開している。
HPLCマップを用いた糖鎖構造解析は、複雑な糖鎖構造を異性体を識別しつつ定量的に解析することが可能であり、ヒューマングライコームプロジェクトにおいても重要な役割を担っている。本記事では、HPLCマップの基本原理と応用、特に質量分析との連携やデータベース「GALAXY」の活用による糖鎖構造の同定方法について紹介する。この手法は、糖鎖研究の加速やヒト糖鎖地図の精密化に大きく貢献する。
糖鎖は、構造の多様性と複雑性さらには不均一性により、他の生体高分子にはない独自の解析上の課題を抱えている。ヒューマングライコームプロジェクトをはじめとするハイスループット型のグライコーム研究においては、質量分析(MS)技術が構造解析の中核を担っている。MS法では、微量の試料を用いて高感度・高精度での質量決定が可能であり、タンデムMSによって構成単糖(HexやHexNAcなど)の組成、並び順、さらにはタンパク質上の修飾部位を迅速に決定できる。しかしながら、糖鎖の生体機能は、構成単糖の連結様式、分岐構造、立体異性、末端修飾といった微細な構造差に依存しており、標準的なMS法ではこれらの識別は困難である。特に、同一の単糖組成を持つ構造異性体の分離と同定は、糖鎖構造解析における大きなボトルネックとなっている。
これに対し、高橋らが開発した高速液体クロマトグラフィー(HPLC)を用いた糖鎖解析手法は、溶出時間の差に基づいて構造異性体の識別が可能であり、構造推定や比較解析において重要な役割を果たしている。この特性を活かした「HPLCマップ」は、糖鎖構造を同定するための強力なツールである1-4。さらに、MSや酵素処理と組み合わせることで、糖鎖の化学構造を高精度に決定できる。本稿では、HPLCマップを用いた糖鎖解析法の原理と応用について概説する。
HPLCマップ解析では、糖鎖の還元末端を蛍光試薬(2-aminopyridine, PA)でラベル化し、高感度検出が可能な状態でHPLCにより分離する。得られた溶出時間のデータと既知のデータとを比較することで、構造を同定する(図 1)。HPLCの溶出時間は糖鎖の構造に強く依存し、分岐様式、末端修飾(フコシル化、シアリル化、ガラクトシル化など)、リン酸化・硫酸化の有無、蛍光標識ラベルとの相互作用といった要因が分離挙動に影響を与える。この特性により、溶出時間に基づく「マッピング」は、糖鎖の構造情報を視覚的かつ定量的に把握するための強力な手法となる。
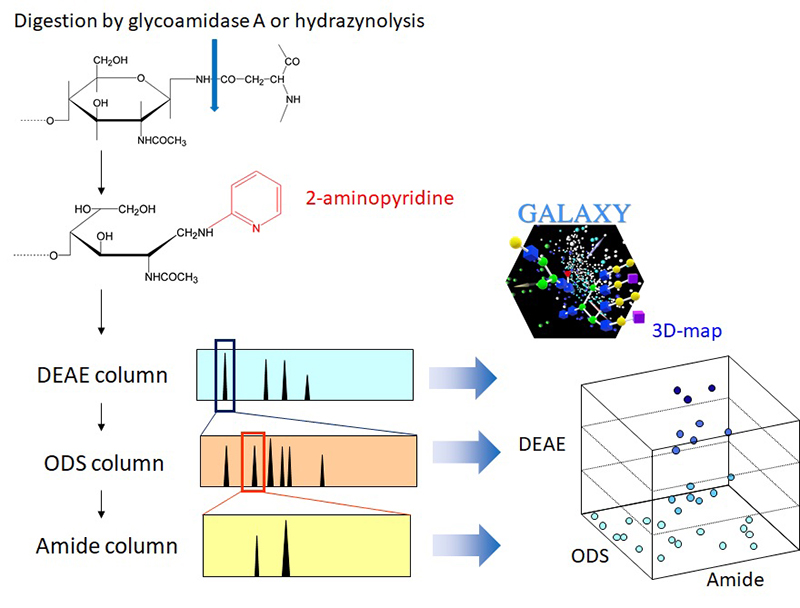
HPLCマップにおいては、陰イオン交換、逆相、順相の各カラムによる分離モードが用いられている。陰イオン交換HPLCには diethyl aminoethyl(DEAE)カラムが利用されており、シアル酸、硫酸、グルクロン酸を含む酸性糖鎖を負電荷数に応じて保持することで分離を可能としている。
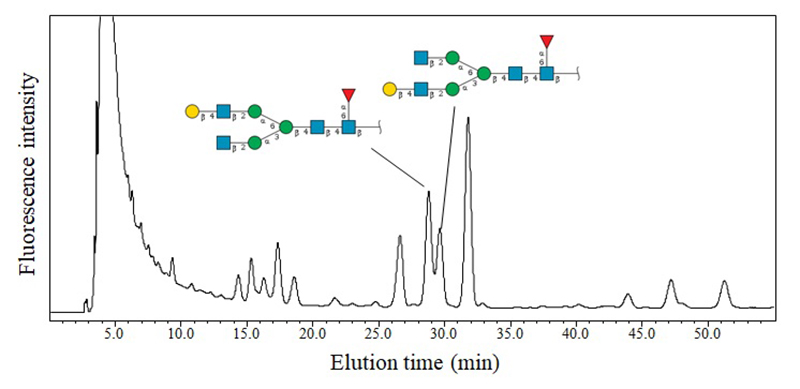
逆相HPLCではoctadecyl silica(ODS)カラムを利用し、糖鎖ごとの微妙な疎水性の違いとカラムの疎水性充填剤との相互作用を利用して分離を行う。溶出条件を最適化することにより、MSでは同定が困難な異性体の識別が可能であり、混在する異性体を分離して定量することができる。図 2は、血清糖タンパク質由来のN型糖鎖プロファイルであるが、ガラクトース残基の結合位置が異なる異性体を分離分析することが可能となっている。
順相HPLCはアミドカラムを利用し、糖鎖の親水性に基づいて分離する。糖鎖の分子量や水酸基数が多い糖鎖ほど溶出時間が長くなる傾向にあり、溶出時間と質量の間に高い相関が得られる。これにより、MSと併用することで、マッピングのスループット性の向上が期待できる。
HPLCマップのデータと実測値を比較するためには、溶出時間を標準化する必要がある。この際、既知の標準糖鎖混合物を基準として算出されるGU(Glucose Unit)値が用いられる。これにより、異なる測定日や装置間、さらには異なる研究機関間でも溶出時間データの比較が可能となる。
また、このマッピング法は、既知構造の同定にとどまらず、未知構造の探索にも対応可能である。溶出時間の差異や酵素処理後の挙動変化を利用して、未知糖鎖の構造的特徴を推定することができ、さらにMSデータと組み合わせることで構造決定の精度が向上する。
HPLCマップ法は蛍光検出によるため、MSに比して定量性が高い。また、HPLCによる分取が可能であり、構造同定した糖鎖をそのまま収集することができる。これにより構築される糖鎖ライブラリーは糖鎖の構造機能解析のための有用なツールとなる5。こうしたこともHPLCマップ法の重要な特長である。
現在、600種以上の既知糖鎖のHPLCデータを収録したデータベースが整備されており、Webアプリケーション「GALAXY」として利用可能である6,7。GALAXYでは、カラムの溶出時間および質量の実測値を入力することで、想定される構造候補を提示する機能を提供している。また、各単糖ユニットの溶出に与える影響(Unit Contribution)をもとに、未知糖鎖のマップ上の出現位置の予測にも活用されている。GALAXYはGlyCosmos Portalと連携しているため、他の外部データベースとのリンクも可能であり8、同定した糖鎖の3次元構造やそれらを含む糖タンパク質に関する情報にもアクセスできるようになっている。
MSによるハイスループット解析では、構成単糖の組成や並び順の情報は得られるが、化学構造を一義的に決定することは難しい。N型糖鎖については、こうした情報が与えられれば生合成経路に関する知識に基づいて大まかな構造を予測することはできよう。しかしながら、シアル酸の結合様式(α2,3かα2,6か)や、ガラクトースの結合位置(β1,3またはβ1,4)といった構成単糖の連結様式の差異を厳密に決定することは一般的には難しい。シアル酸残基については、その結合様式の違いに基づいた反応性の差を利用したSALSA法(Sialic Acid Linkage-Specific Alkylamidation)などにより識別が可能となっている9。また、MSの多段階フラグメンテーション(MSn)を用いて異性体を識別するアプローチも存在するが10、混合試料中の異性体を扱う場合には解釈が煩雑になり、スループット性が著しく低下するという問題がある。こうした課題に対し、HPLCマップ法はMSを補完するツールとして有効である。
一方で、MS法を併用することで、HPLCマップ法の解析精度や信頼性は大幅に向上させることができる。たとえば、アミドカラムでの溶出時間データを質量情報に置き換えることで、構造異性体の同定精度を向上させつつ、スループット性も確保できる。また、広く利用されているLC-MSシステムのように、HPLCマップ法で用いるODSカラムの出口にMS検出器を接続すれば、各ピークに対応する糖鎖の質量および断片構造情報を同時に取得することが可能となる。
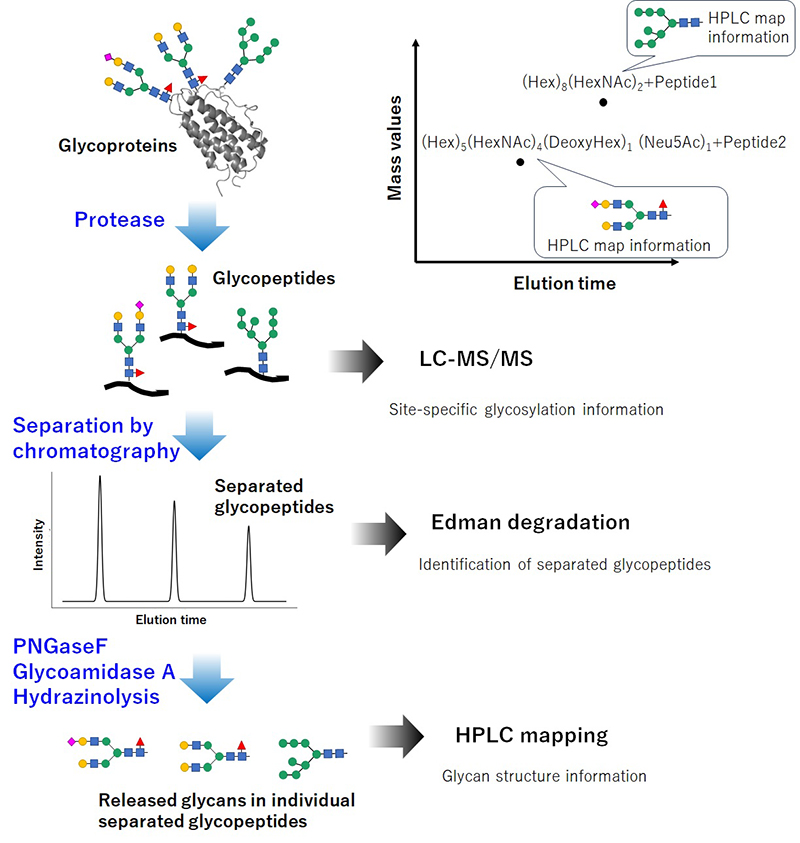
ヒューマングライコームプロジェクトでは、ハイスループット解析によって得られた組成レベルの情報に、HPLCマップに基づく化学構造の情報を統合するアプローチが導入されている(図 3)。複数のN型糖鎖修飾部位を有する糖タンパク質を対象にLC-MS解析を行うことで、それぞれの部位に結合している糖鎖の単糖組成を知ることできる。さらに、それぞれの修飾部位を含む糖ペプチドごとに分離したうえで、HPLCマップ法を適用することで、修飾部位ごとの糖鎖構造を明確に記述することが可能となる。
HPLCマップを用いた糖鎖構造解析は、高感度・高分離能・定量性に加え、視覚的・直感的なデータ解釈を可能とする点で、糖鎖研究に広く用いられている。本手法は、未知の糖鎖構造の探索、定量的比較、バイオ医薬品の品質評価、疾患マーカーの発見など、幅広い分野で活用されている。ヒューマングライコームプロジェクトにおいても、HPLCマップ法はMSを補完する重要な役割を演じている。
HPLCマップ法の基盤となるデータベースは拡充が進んでおり、試料調製から計測までのプロセスがオートメーション化されれば、さらに多くのデータが蓄積されると期待される。これらの大規模データを教師として機械学習を活用すれば、糖鎖構造の同定や未知糖鎖の構造予測が飛躍的に進展するであろう。その成果は、ヒト糖鎖地図の精密化にとどまらず、自然界に存在する糖鎖の網羅的な構造解析とライブラリー化を促し、さらには非天然型糖鎖の構造解析へと応用範囲を広げる可能性を秘めている。