
氏名:浦島 匡
国立大学法人帯広畜産大学、農学博士
1986年東北大学博士取得後、帯広畜産大学畜産学部酪農化学教室に赴任、ミルクオリゴ糖の研究に従事。1991年文部省在外研究員としてシドニー大学生化学部門にて、Dr. Michael Messerの下でタマーワラビー(有袋類)の乳腺における糖転移酵素の研究に従事。以後、Messerと共同で単孔類、有袋類、いくつかの種の有胎盤類のミルクオリゴ糖の化学構造の比較研究を行う。哺乳類への進化過程で、現在の乳成分がどのように獲得されたかに興味をもち、とくにミルクタンパク質αラクトアルブミンの獲得がミルクオリゴ糖とラクトースの発生と生理的意義の変化にどのように影響を及ぼしたかの探索を生涯の研究テーマにしている。2003年~2022年、帯広畜産大学教授。現在、名誉教授。日本糖鎖科学コンソーシアム幹事。

氏名:福田 健二
国立大学法人帯広畜産大学 教授
2002年北海道大学大学院農学研究科にて博士号(農学)取得。学位取得後、2004年までカールスバーグ研究所とデンマーク工科大学において博士研究員。2005年国立大学法人帯広畜産大学助教、2010年准教授、2021年より現職。「ワンヘルス」の概念の下、家畜乳と発酵乳製品に含まれるオリゴ糖やペプチドなどの生物活性物質の探索、乳酸菌が生産する多糖類の特性評価や応用について研究している。

北岡 本光
国立大学法人新潟大学農学部、博士(農学)
1985年東京大学工学部反応化学科卒業、民間企業を経て1993年論文博士。1995-1998年アイオワ州立大学博士研究員としてデキストラン合成酵素の反応機構を研究。1998-2019年農研機構食品研究部門にて酵素を利用したオリゴ糖の実用的生産技術の開発を中心にした研究に従事。2019年4月より新潟大学教授。

伏信 進矢
東京大学大学院農学生命科学研究科 教授
1994年東京大学農学部農芸化学科卒業。1996年東京大学大学院農学生命科学研究科応用生命工学専攻修士課程修了。1997年東京大学大学院農学生命科学研究科助手。1999年博士号取得(東京大学)。2006年米国アイオワ州立大学Visiting Scientist。2011年東京大学大学院農学生命科学研究科准教授。2012年同大学院教授。
各種の酵素の構造と機能に関する研究を行っている。特に、糖質関連酵素に関する研究が中心となっている。

氏名:片山 高嶺
京都大学大学院生命科学研究科教授
京都大学農学部食品工学科を卒業後、同大学院に進学し、熊谷英彦先生の下で学位を取得。応用微生物学分野を専門とする。同研究室で3年間博士研究員として在籍した後に、京都大学大学院生命科学研究科統合生命科学専攻・山本憲二教授研究室の助手に着任し、糖質関連酵素に関する研究をスタートさせる。ビフィズス菌がヒト由来の糖質に作用する酵素を有していることを見出し、ヒトと腸内細菌の共生に着目する。母乳栄養児におけるビフィズスフローラ形成機構の解明に貢献した。
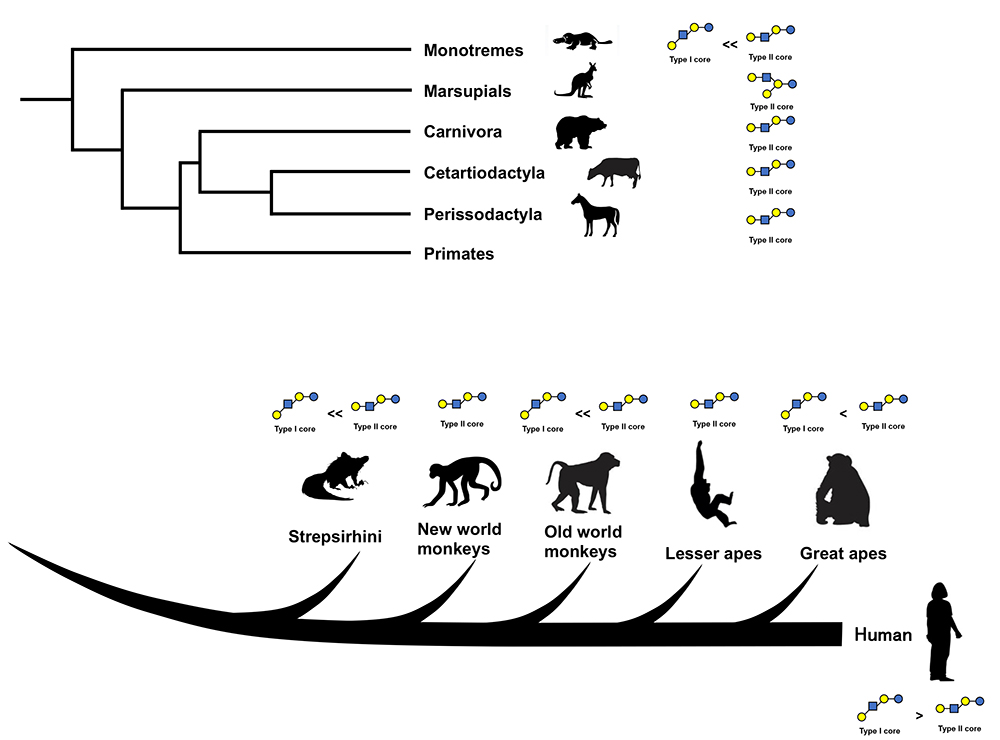
2011年5月にコペンハーゲンで開催されたGlycom A/S社(現在はDSM社)主催の第1回human milk oligosaccharide, glycobiology symposiumにおいて、浦島は“The predominance of type I oligosaccharides is a feature specific to human breast milk”と題する講演を行なった。この仮説は、浦島らが解析してきた霊長目、食肉目、鯨偶蹄目、長鼻目、有袋類、単孔類のミルクオリゴ糖構造とヒトミルクオリゴ糖(HMOs)構造のプロファイルを比較・観察した結果提案されたが1、当時はHMOsにおいてタイプ2型のラクト-N-ネオテトラオース(LNnT)が最優先であるといくつかの論文や総説論文に記載されていて2,3、それへの認知は十分に広がらなかった。一方、LNnTが最大であるという根拠となった高速液体クロマトグラフィー(HPLC)によるHMOsのプロファイルでは、LNnTとラクト-N-テトラオース(LNT, タイプ1型)のピークの相互分離が不十分であった2。200種類のHMOsの中で、Thurlら(2010)によるその後発表された優先的なHMOsに対する定量データでは、LNTとLNnT、およびLNTまたLNnTをコアとするHMOsで、分泌型ドナーで前者の方が4~5倍高く、非分泌型ドナーではその差はさらに大きかった4。同論文に対する被引用数は450を超えており、一般的に認知されているとみなしてよい。HMOs間でのタイプ1型OS優先性は、現在では共通のコンセンサスとなっている。
ヒト以外の哺乳動物のミルクでは、タイプ1型ミルクオリゴ糖(ミルクOS)とタイプ2型ミルクOSの割合はどうであろうか?単孔類(ハリモグラ、カモノハシ)では、カモノハシのミルクでLNTをコアとするタイプ1型OSが1例のみ報告されている一方、数種類のLNnTやラクト-N-ネオヘキサオース(LNnH)をコアとするタイプ2型OSが同定されており5,6,7、定性的にも定量的にもタイプ2型OSが優先的である。有袋類(タマーワラビー、アカカンガルー、ブラッシュテイルポッサム、ウオンバット、コアラ、イースタンクオール、タイガークオール)では、ラクトースコア以外のミルクOSはもっぱらラクト-N-ノボペンタオース I (novo LNP-I、タイプ2型)などをコアとするタイプ2型であった5,6,7。多くの哺乳動物のミルクの糖質において、ラクトースはミルクOSよりも優先的であるが、単孔類、有袋類の他、食肉目イヌ亜目の種ではミルクOSの方が優先的である5,6,7,8。ヒトやゾウ(アジアゾウ、アフリカゾウ)のようにラクトースが優先的であるものの、ミルクOSが糖質の20 %~40 %を占める種もいる6,9,10。従来ミルクオリゴ糖の構造は、ミルクから各OSを分離・精製してから核磁気共鳴装置(NMR)のような機器分析によって決定されていたが、ミルクOSの豊富なミルクに含まれるOSの構造解析は比較的容易であった。浦島らはアメリカクロクマ、ホッキョクグマなどのクマ科動物、アライグマ、ミンク、スカンク、ズキンアザラシなどのアザラシ科動物、アジアゾウ、アフリカゾウ、ミンククジラなどのミルクにおいて、LNnT, LNnH, パラLNnHなどのタイプ2型OSをコアとする構造を同定したが、タイプ1型OSをコアとする構造は発見できなかった5,6,11,12。一方、HPLCなどによって分離し、NMRを使用して構造解析する方法では、NMRの検出感度の低さが原因で、優先性が低いOSまでは構造決定に至っていない。上のような種のミルクにタイプ1型OSは、優先性は低いながらも含まれている可能性は否定できない。実際にハイスループットなグライコミクス方法によって、イエイヌ、イエネコとライオンのミルクにタイプ1型OSが発見されている13,14。
Marinoら(2010)はウシミルクOSを、Albrechtら(2014)は、ウシ、ヤギ、ヒツジ、ラクダ、ウマ、ブタのミルクOSを、蛍光標識後の高速液体クロマトグラフィー(HLPC)―質量分析(MS)とエキソグリコシダーゼ消化の組み合わせによって構造決定した15,16。かれらの用いた方法では低い濃度のOSまで網羅的に構造決定が可能である。構造決定されたミルクOSの中にラクトースをコアとする構造以外は、LNnT, LNnH, novo LNP-I、またイソLNnTなど、タイプ2型OSをコアとするOSは発見されたが、タイプ1型OSをコアとするOSは発見されなかった。Remorozaら(2020)による分析でもウシとバッファローのミルクにタイプ1型OSは発見されなかった14。家畜動物のミルクに、いくつかタイプ1型OSを発見した例はある。例えば、蛍光標識後のキャピラリー電気泳動またはHPLCによる分離とMSの組み合わせの技術によって、ヤギミルクにLNTとLNH(ラクト-N-ヘキサオース、タイプ1型構造を含む)が14、ウマミルクにLNT、LNH, シアリルラクト-N-テトラオース a (LST-a), ジシアリルラクト-N-テトラオース(DSLNT)が17,18、ブタミルクにLNT, ラクト-N-フコペンタオースII (LNFP-II), LNH, ラクト-N-ジフコヘキサオース(LNDFH), LST-a, LST-b, シアリル(S)-LNFP-II, S-LNH, フコシルシアリル(FS)-LNHが報告された19。家畜ミルクにタイプ1型OSを発見しなかったAlbrechtら16とそれらを発見したDifilippoら18,19の結果の違いが、特にウマとブタミルクOSに関して注目される。これには品種間差また個体間差の違いも予想されるが、分析技術の違いによる可能性もある。MS単独での構造解析ではGalβ1-3GlcNAc単位とGalβ1-4GlcNAc単位や、Galβ1-3(Fucα1-4)GlcNAc単位とGalβ1-4(Fucα1-3)GlcNAc単位などがフラグメンテーションパターンの違いで区別しにくく、熟練した観察が求められる。基質特異性の異なるβ-ガラクトシダーゼを使用した酵素消化を分析に加えることも必要であろう。
これらの家畜種のミルクオリゴ糖の中で、ラクトースをコアとするもの以外では、タイプ2型OSしか発見されないか、またはタイプ2型OSがタイプ1 型OSよりも優先的であることは疑いない。
ヒトに近縁な種の霊長目では、タイプ2型OSとともにタイプ1型OSも存在する。チンパンジーミルクにはLNT(タイプ1型)とLNnT(タイプ2型)が1:1の割合で含まれていたが、LNFP-III(タイプ2型)は検出される一方、タイプ1型のLNFP-IやLNFP-IIは検出されなかった20。ボノボではLNT、LNFP-IとLNFP-IIIが発見され、LNFP-I vs LNFP-III割合は1:4であったので20。同様にタイプ2型優先的といえる。ゴリラではLNnTは発見されたが、LNTは発見されなかった20。オランウータンではLNT、LNnT、LNFP-III、ジフコシル(DF)LNnH(タイプ2型)、LST-b(タイプ1型)、LST-c(タイプ2型)、FSLNnH(タイプ2型)が発見されたが、ゲル濾過やHPLCのピーク強度からタイプ2型の方が優先的であった20。カンムリテナガザルではタイプ2型のLNnT、LNnH、LST-c、SLNnHは発見されたが、タイプ1型OSは検出できなかった20。このように、類人猿のミルクOSではタイプ2型がタイプ1型よりも優先的であった。
旧世界ザル(アカゲマカク、トクマカク、マントヒヒ)と新世界ザル(フサオマキザル、ボリビアリスザル、マントホエザル)のミルクでは、LNnT、LNFP-III、novo LNP-I、LNnH、DFLNnH、LST-c、SLNnHなどのタイプ2型OSは発見されたものの、タイプ1型OSは検出できなかった21。曲鼻猿(原猿亜目)(オオガラゴ、アイアイ、コレルシファカ、マングースキツネザル)のミルクではアイアイのみにタイプ1型OSのLNTとLNFP-IIが発見されたが、タイプ2型OSの方が優先的であった22。他種のミルクではタイプ2型OSしか発見されなかった22。
このように霊長目のミルクには、ウシ、ヒツジ、ラクダのような家畜偶蹄目とは異なってタイプ2型とともにタイプ1型OSも含まれる。けれどもタイプ2型OSの方が優先的であったので、タイプ1型ミルクOS優先性はヒト固有に獲得された性質と考えて良いであろう。なお、上のような霊長目のミルクオリゴ糖解析は、OS分離後の1H-NMRによる解析をベースとしており、マイナーなOSの同定までには至っていない。HPLC-MSのような少量の試料で可能な分析方法では旧世界ザル、新世界ザル、曲鼻猿のミルクにタイプ1型OSが検出できるかもしれない。実際にHPLC-MSをベースとするグライコミクス方法では、チンパンジー、ゴリラ、カンムリテナガザル、アカゲマカク、コモンマーモセット、ゴールデンライオンタマリンのミルクに多くの種類のタイプ1型OSと、タイプ2型OSが発見されている23。これまでに分析されたサンプル数が少なく、もっと多くのミルクを採集して分析を進めるべきであろう。
このように、ミルクオリゴ糖のプロファイルと哺乳動物の系統とは一定の相関が認められた(図 1)。一方でそのような法則性と全く異なる分析結果がKunzらによってアジアゾウ24、Warrenらによって12種(ゴリラ、オランウータン、チンパンジー、ボノボ、ハイイログマ、クロクマ、イヌ、キノボリカンガルー、キリン、クロサイ、フロリダマナティー、バンドウイルカ)のミルクOSプロファイルから得られた25。多くのタイプ1型OSがこれらのミルクに同定されている。これらはいずれもミルクOS構造についての情報が少なかった初期の研究で、構造の決定は主にHMOsとのHPLC保持時間の比較によって行われている。当時、動物のミルクOSはHMOsと大きくは違わないというバイアスが入っていたことが懸念される。多くの種のミルクOSを分離・精製し、NMRなどによって構造解析すると、β1-3ガラクトースの連鎖構造、非還元末端でのA抗原[GalNAcα1-3(Fucα1-2)Gal]やB抗原[Galα1-3(Fucα1-2)Gal]、α-Galエピトープ(Galα1-3Galβ1-4GlcNAc)、novo LNP-Iをコアとするオリゴ糖などHMOsには見られない構造が広範囲に発見され、ヒトと他種のミルクオリゴ糖構造には大きな違いがあった。現在、グライコミクス手法による網羅的な解析が主流となっているが、ミルクOSの初期の研究において、個々のミルクOSを分離し、一つ一つ解析する構造決定が有意義であったことは間違いない。一方でそのような解析では一定量の乳試料の採集を必要とするので、大量に試料採集できる家畜以外の動物の乳を集める困難はあった。
最近D. BojarらのグループによるLC-MS/MS分析とエキソグリコシダーゼ消化を組み合わせたグライコーム分析によって、9の哺乳動物種(アルパカ、ベルーガ、クロサイ、バンドウイルカ、インパラ、ロストグエノン、コビトカバ、家畜ヒツジ、スジイルカ)のミルクから、100の新規構造を含む400ものミルクOSの構造が決定され、構造多様性についての情報は飛躍的に拡大した26。スジイルカと家畜ヒツジ以外のミルクにGalNAcβ1-4GlcNAc(LacdiNAc)を含むOSが、インパラ、バンドウイルカ、スジイルカ、ヒツジ、クロサイにグルクロン酸(GlcA)を含むミルクOSの発見されたことが注目される。これまでにLacdiNAcを含むミルクOSは、ウシに遊離のLacdiNAcが1例発見されているのみである15。LNnTやLNnHへの類似構造としてLdiNnTおよびLdiNnHコアを含むミルクOSがインパラ、コビトカバに発見された。LacdiNAcを含むミルクOSは鯨偶蹄目のミルクに拡がっている他に、霊長目のロストグエノン(旧世界ザル)にも発見されたことが注目される。ラクトースの生合成において、β4ガラクトシルトランスフェラーゼ1(β4GalT1)とミルクタンパク質α-ラクトアルブミン(α-LA)の会合によって、β4GalT1のアクセプターがGlcNAcからGlcに代わるが27、in vitroの試験では両タンパク質の会合によって前者のドナーは、UDP-GalからUDP-GalNAcにも変わりうる27,28。これらのミルクOSに発見されたLacdiNAc構造単位は、このようなα-LAとの会合によるβ4GalT1分子の基質のよろめきによって生合成されたかもしれない。
Sdaエピトープ[Neu5Acα2-3(GalNAcβ1-4)Gal]を含むミルクOSが、ベルーガ、バンドウイルカ、クロサイに発見された26。これまでに同エピトープを含むミルクOSは最小構造のNeu5Acα2-3(GalNAcβ1-4)Galβ1-4Glc(GM2 tetrasaccharideと命名)のみが、バンドウイルカ29、アカゲマカク21、キリン30に発見されていたが、新たにLNnT, LNnH, LdiNnT, LdiNnHのようなコア骨格にも付加した構造が発見された。浦島らによるミルクから分離・精製し、主にNMR手法によって構造決定する方法では最小の4糖しか発見できなかったが、微量の試料で解析できるグライコーム技術の進歩により新たな境地が開かれた。MSnによる構造特異的なフラグメンテーションパターンの経験則への知識の蓄積と、基質特異的なエキソグリコシダーゼの使用からもたらされる情報によって、構造解析技術は飛躍的に進歩した。従来の解析で見落とされてきた微量のOSも含めて解析できるようになったことから、多くの種でより精密な多様性をもったミルクOSのプロファイルが明らかになっていくであろう。ミルクOSの構造解析技術の進歩は、HMOsの構造解析方法の歴史的な変遷とも共通している。HMOsは、1970年代までは単糖組成分析、メチル化分析、エキソグリコシダーゼ消化の組み合わせによる古典的な技術によって、1980年〜2000年は一次元、二次元NMRをベースとした方法によって構造決定されたが、NMRによる構造決定もChaiら(2005)によるラクト-N-デカオース(LND)とフコシルラクト-N-デカオースI (F-LND-I)の発見31を最後として、それ以降はMSn分析をベースとする方法によって構造決定されている32,33,34,35,36。
Jinらの研究によってミルクOSの多様な構造情報は飛躍的に拡大したが26、タイプ1型OSとタイプ2型OSの哺乳類種間分布については、従来の観察と大きくは違わない(図 1)。霊長目旧世界ザル:ロストグエノン以外では、タイプ1型ミルクOSはほとんど発見されなかった。ロストグエノンでは、ラクトースについで優先的なミルクOSは、LST-b(タイプ1型)であった。Gotoら(2010)は近縁種のアカゲマカクとマントヒヒミルクにLST-c(タイプ2型)を発見したが21、LST-bは検出しなかった。LST-bの存在は旧世界ザルの中でもロストグエノンにユニークであるかもしれない。
構造決定されているミルクOSの網羅的なデータベースが作成され(MilkOligo DB)37、哺乳動物種の系統とミルクOS構造の相関と進化、可能な生合成経路についても予想されている37,38。Tomesら(2023)によると、有袋類のミルクOSは、他種のミルクOSとは独自にグルーピングされるように進化をしていることが指摘される一方38、Warrenら(2001)によるキノボリカンガルーのミルクOSデータ25は、相互に相同性をもった有袋類ミルクOSグループとは大きく乖離している。上述したように、Warrenらによる12種のミルクOSデータは、ミルクOSの系統進化についての考察には加えない方が都合がよい。
2011年の第1回HMOsグライコバイオロジーシンポジウム以降、Albrechtら(2014)によるウシ、ヤギ、ヒツジ、ラクダ、ウマ、ブタのミルクOS解析16、Remorozaら(2020)によるウシ、スイギュウ、ヤギ、ライオンのミルクOS解析14、そしてJinら(2023) による9の哺乳動物種のミルクOS解析26によって、ヒト以外にもミルクOS構造情報は飛躍的に拡大した。それによって、タイプ1型ミルクOS優先性はヒト固有に特徴的という仮説はさらに支持されたようだ。
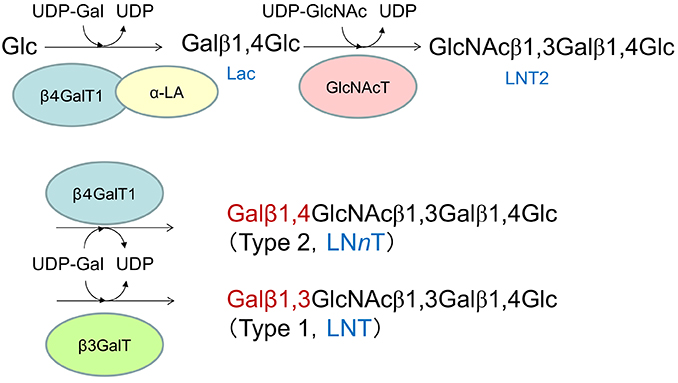
タイプ1型および2型OSは、それぞれのコア4糖(LNT, LNnT)を出発物質として生合成される。これらのコア4糖は、ラクトースの非還元末端Gal残基の3位にGlcNAcが転移して生じるラクト-N-トリオースII (LNT2)に、異なるガラクトース転移酵素によりGalが転移することによって生成する(図 2)。このため、LNTおよびLNnTの生合成は、LNT2およびUDP-Galを基質とした2種のガラクトース転移酵素による競争反応となる。ここで、β4GalT1はラクトースの生合成に関わる酵素と同じであるため、ラクトースを主要糖とする動物種の乳腺には多量に発現していると考えられる。そのため、タイプ1型優勢のミルクOS組成を得るためには、β4GalT1よりも高いβ3GalT活性が必要となる(図 2)
ヒトはウシに比べてミルクオリゴ糖の濃度は100倍くらい高く、種類も豊富である。HMOsは、種類豊富で高濃度そしてタイプ1型優先性という方向に進化した。現在HMOsの生理機能に対する研究論文も増え、有用性腸内細菌ビフィズス菌に対する増殖・定着促進、病原性ウィルスや細菌への感染防御、免疫調整、壊死性腸炎の予防、脳神経系の発達促進などの機能性が注目されている39。HMOsによるこれらの機能には、乳児の腸内ビフィズス菌の代謝産物によるメカニズムも予想される。この15年間で主に日本とカリフォルニア大学デービス校の研究グループによって、タイプ2型とともにタイプ1型HMOsに対するヒト乳児型ビフィズス菌の代謝経路が発見された。ヒトミルクにおけるタイプ1型HMOs優先性への進化とビフィズス菌によるタイプ1型HMOs代謝経路の獲得によって、ヒトとビフィズス菌の共生進化がもたらされ、そのことで母乳栄養児の健全な発達に重要な役割をもつようになった。乳児型ビフィズス菌によるタイプ1型HMOsへの資化性の獲得に至ったストーリーについて簡単に説明する。
母乳栄養乳児腸管でのビフィズス菌の優先増殖は20世紀初頭から知られており、1960年ころには母乳に含まれるHMOsが増殖因子として作用していると推定されていた。1999年にタイプ1型OSの末端構造二糖であるラクト-N-ビオースI(LNB)を選択的に加リン酸分解する酵素である、β-1,3-ガラクトシル-N-アセチルヘキソサミンホスホリラーゼ(GLNBP)がBifidobacterium bifidum菌体内に発見された40ことにより、ビフィズス菌のHMO代謝系の理解が急速に進んだ。GLNBP遺伝子が、複数のビフィズス菌種から同定されたことから、ビフィズス菌はタイプ1型OSの末端からLNBを遊離して選択的に資化することにより優先増殖を得ているとするLNB仮説が提唱された(図 3右)41。GLNBP遺伝子は、主要な乳児定着型ビフィズス菌種に広く分布するが、それ以外のビフィズス菌種にはほとんど分布していなかった42。また、LNB資化性ビフィズス菌種は、GLNBP遺伝子保持菌種にほぼ限定されていた42。LNB仮説に従い、タイプ1型OSからLNBを遊離するのに必要な菌体外酵素系(フコシダーゼ、シアリラーゼ、ラクト-N-ビオシダーゼを含む)がB. bifidumから同定された43。しかしながら、ゲノム解析の進歩によりこのような菌体外酵素系を持つビフィズス菌種はB. bifidumのみであることが示された44。
一連の菌体外酵素系としてフコシダーゼ、シアリダーゼ、およびラクト-N-ビオシダーゼのセットを保持する菌種はB. bifidumのみであるが、LNTに加えてLNFP-I (Fucα1-2Galβ1-3GlcNAcβ1-3Galβ1-4Glc)やLST-a (Neu5Acα2-3Galβ1-3GlcNAcβ1-3Galβ1-4Glc)にも作用可能な菌体外ラクト-N-ビオシダーゼ(GH136)活性が、Bifidobacterium longum subsp. longum (B. longum) の1/3~1/2程度の株に発見されている45。同酵素やその発現のためのシャペロンタンパク質の獲得も、タイプ1 型HMOs資化性へのビフィドバクテリウムの適応の一部であると考えられる。また、同じニッチに生息するB. bifidumとB. longumにおいて、全くアミノ酸配列の異なるアイソザイムとしてラクト-N-ビオシダーゼを獲得した点でも興味深い。
タイプ1型HMOs資化性への適応において、Bifidobacterium longum subsp. infantis (B. infantis)によるHMOs代謝も重要な視点である。B. infantisのゲノムにおいては、HMOsの代謝系に関わるトランスポーターやグリコシダーゼの遺伝子の集まった43 キロベースペアに及ぶ領域(HMOクラスター1と命名されている)が発見されているが46、HMOクラスター1内にコードされるβ-ガラクトシダーゼには、タイプ2型OSに対する基質特異性はあるもののタイプ1型OSへの加水分解能は保持していない。一方、吉田らは、HMOクラスタ―1外の遺伝子座にコードされるβ-ガラクトシダーゼにタイプ1型OSへの分解能があることを発見した47。B. infantisは3種のGH42β-ガラクトシダーゼを保持しているが、そのうちの一つBga42Aにはタイプ1型OSのLNT以外に、Galβ1-3Galβ1-3Galβ1-4Glcに対する加水分解能も観察された48。このことは、本来Galβ1-3Gal単位に対する親和性をもつβ-ガラクトシダーゼが、LNTのようなタイプ1型OSに対しても加水分解能をもつように適応した可能性を示している(図 3左)。

一方、乳児型ではないビフィドバクテリウム菌種のBifidobacterium animalis subsp. lactisBl-04株由来のGH42A β-ガラクトシダーゼはGalβ1-3Galに対する加水分解能をもつものの、LNB、LNT、Galβ1-3GalNAc (GNB)、またGalβ1-3Galβ1-4GlcとGalβ1-3Galβ1-3Galβ1-4Glcを加水分解できない49。B. infantisのBga42AによるLNTへの加水分解能には、切断位置に隣接するバルキーな置換基 (NAc)の受容と、3糖以上のオリゴ糖に対する受容の2つが獲得されなければならず、どのような分子進化があったのか興味のもたれる点である。
このようにB. bifidumやB. infantisによって獲得されたタイプ1 型HMOsへの資化能は、菌体外LNBの利用などによって、Bifidobacterium breveやB. longumを含めた腸内ビフィズスフローラの形成に重要な意義を持ったに違いない50,51。
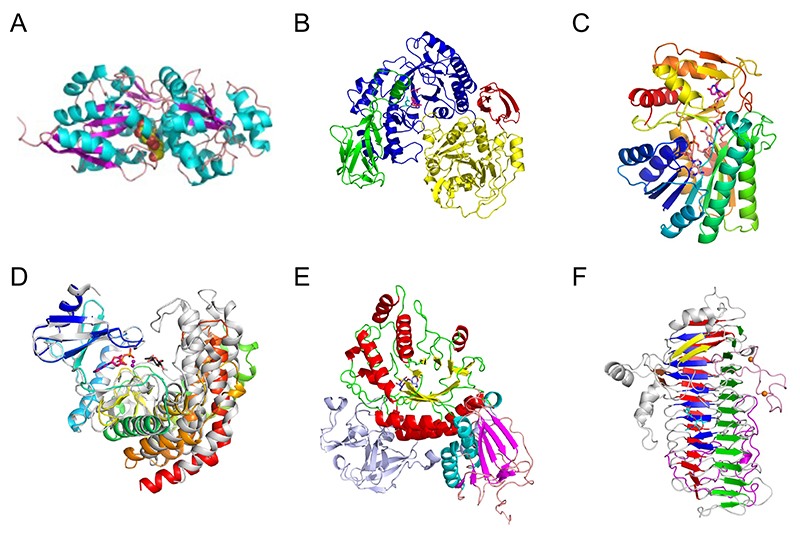
タンパク質の立体構造は異なるファミリーの間でも保存性が見られることから、どのような分子進化を経てきたかを考える上での手がかりになる。ビフィズス菌のタイプ1型OSの特異的代謝経路に関わる酵素・タンパク質において、最初に立体構造が報告されたのが、オリゴ糖を菌体内に取り込むABCトランスポーターの基質結合タンパク質である(図 4A)52。これはB. longum由来のタンパク質であり、GNBもLNBと同様に強く認識するためにGNB/LNB結合タンパク質(GL-BP)と呼ばれている。GL-BPは二糖であるLNBの全体を極めて強く認識しており、このタンパク質がタイプ1型OSの菌体内への取り込みに適応して分子進化してきたことが推察される。糖質の基質結合タンパク質の全体構造は、有名なマルトデキストリン結合タンパク質(MBP)も含めてどれもよく似ており、N末端とC末端の2つの大きなドメイン単位からなっていることから、これらは基質結合部位周辺の変異により、多様な糖質の取り込みを担うようになってきたのだろう。B. longumのGL-BPは四糖であるLNTに対する結合は弱いが、B. infantisのホモログではLNTもLNBと同様に強く結合することがわかっている。ビフィズス菌の菌種ごとに、菌体外の分解酵素の存在に合わせてトランスポーターの特異性も微調整が行われている様子がうかがえる。
続いて、菌体内でLNBを加リン酸分解するGLNBPの立体構造も報告された(図 4B)53。GLNBPは基質結合により触媒ドメインの大きな構造変化が起こる。GLNBPはGH112に属するが、その触媒ドメインは(β/α)8バレルであり、GH14のβ-アミラーゼと似ているため、分子進化的関連性が推察される。ただし、タイプ1型OSとβ-アミラーゼの基質であるα-1,4-グルカン(澱粉)はかなり化学構造が異なり、反応機構も加水分解と加リン酸分解で異なるため、触媒ドメインの動きを伴うやり方で基質認識と触媒反応(リン酸結合)に対応しているのではないかと考えられる。GLNBPの反応産物であるガラクトース 1-リン酸(Gal-1P)とGlcNAcをさらに代謝する下流の酵素は、ガラクトース代謝を行ういわゆるLeloir経路の酵素と似たような酵素が担う。そのうち、UDP-Glc/UDP-GalおよびUDP-GlcNAc/UDP-GalNAcを変換するエピメラーゼ(GalE)は大腸菌やヒトなどの酵素と配列も構造もよく似ている(図 4C)54。しかし、GlcNAcの1位(アノマー位)にATPからリン酸を転移するアノメリックキナーゼであるN-アセチルヘキソサミンキナーゼ(NahK)や、糖1リン酸(Gal-1PやGlcNAc-1Pなど)とUDP糖の間で糖を転移してGalEの基質を供給する糖1リン酸ウリジリル転移酵素(GalT)は、ビフィズス菌のタイプ1型OS(LNB)代謝経路においては既知のLeloir経路の酵素とは異なる独自のアミノ酸配列を持つ酵素が使われている。特に、B. longumのNahKはプロテインキナーゼやアミノグリコシドの糖転移酵素に立体構造が似ており(図 4D)、これらの酵素から流用されている可能性がある55。
菌体外で四糖以上のタイプ1型OSからLNBを切り出すラクト-N-ビオシダーゼは、B. bifidumではGH20の酵素(LnbB)が用いられている(図 4E)56。GH20の大部分は単糖のGlcNAcまたはGalNAcを切り出すエキソ型酵素(N-アセチルヘキソサミニダーゼ)であるため、これが祖先型酵素であると推測できる。LnbBは二糖を遊離できるようにβ1-3結合したGalを受け入れるポケットを持つように分子進化してきたのだろう。一方、B. longumの持つラクト-N-ビオシダーゼは先述したようにGH136の酵素(LnbX)である。LnbXはこれまで示してきた数々のタイプ1型OSを分解・代謝する酵素・タンパク質に比べても、極めてユニークなβ-ヘリックス構造をとっている(図 4F)57。LnbXの立体構造は既知の酵素との相同性は低く、ファージのテイルスパイクタンパク質とやや似ている。このようなタンパク質には糖鎖結合能を持つものもあり、ビフィズス菌のゲノムに含まれるファージ由来の配列(プロファージ)がヒトとの共生において分子進化や多様性の獲得に影響を与えた可能性も示唆されている58。
リゾチームからの分子進化によるα-ラクトアルブミンの出現によって、ラクトースとともにミルクオリゴ糖の生合成が開始された5。現存の哺乳類の中で形態的に祖先の特徴を残す単孔類のミルクにおいてラクトースよりもミルクオリゴ糖の方が優先的なように、初期の哺乳類では糖質の中でミルクオリゴ糖が優先的なミルクを分泌していたことが予想される5,6,7。これまでにミルクオリゴ糖が研究されている哺乳類の中で、霊長目以外のミルクにはラクトースやガラクトシルラクトースをコアとするミルクオリゴ糖以外に、タイプ2型OSが広範囲に発見されるのに対し、タイプ1型OSは含まれないか、含まれていても痕跡程度の濃度である1。霊長目のミルクではタイプ1型OSとタイプ2型OSが発見されるものの、後者が前者よりも優先的である1,23。一方ヒトのミルクでは、特異的にタイプ1型OSがタイプ2型OSよりも優先的で1,4 ,ミルクオリゴ糖の濃度もウシやヤギよりもはるかに高い。
ヒトミルクに含まれるHMOsは、ヒト乳児の健康に重要な役割を担う腸内細菌ビフィズス菌によって代謝され、ヒトとビフィズス菌との共生をもたらした。ヒト型ビフィズス菌が乳児の腸内で定着するため、母乳に優先的なタイプ1型HMOsへの代謝能の獲得が鍵であった。そのような代謝能は、HMOs代謝に関わるトランスポーターやグリコシダーゼ、ホスホリラーゼの一部に、タイプ1型HMOsやLNBへの親和性の獲得によってもたらされた。
HMOsには母乳栄養児に対して病原性微生物への感染防御、免疫調整、壊死性腸炎予防、脳神経系の発達促進、腸管バリア機能向上などの生理的機能があるが39、機能の一部は腸内細菌とくにビフィズス菌によるHMOs代謝産物によって担われる。未熟な状態で出生するヒト乳児の健康維持にビフィズス菌が重要な働きをもつことは間違いない。近年末梢神経を介した脳腸相関によって、腸内細菌代謝物の脳機能への働きが証明されているが、腸内に定着するビフィズス菌がヒト乳児の脳の発達に大切な役割を持つことも予想される。そうであれば、ミルクオリゴ糖の中でタイプ1型OS優先性の獲得によって、ビフィズス菌との共生が促進され、それがさらにヒトの進化を促したという想像もできる。牛乳を原料として製造される育児用調整乳に、工業的に生産されるようになった少数のHMOs(2’FL, LNnT)が添加されているが、LNT, LNFP-I, LNDFH-I, DSLNTなどのタイプ1型HMOsの工業的な生産と育児用調整乳への添加が期待される。
lacto-N-triose II (LNT2) : GlcNAcβ1-3Galβ1-4Glc
lacto-N-tetraose (LNT) : Galβ1-3GlcNAcβ1-3Galβ1-4Glc
lacto-N-neotetraose (LNnT) : Galβ1-4GlcNAc1-3Galβ1-4Glc
lacto-N-hexaose (LNH) : Galβ1-3GlcNAcβ1-3(Galβ1-4GlcNAcβ1-6)Galβ1-4Glc
lacto-N-neohexaose (LNnH) : Galβ1-4GlcNAcβ1-3(Galβ1-4GlcNAcβ1-6)Galβ1-4Glc
lacto-N-novopentaose I (novo LNP-I) : Galβ1-3(Galβ1-4GlcNAcβ1-6)Galβ1-4Glc
para lacto-N-neohexaose (para LNnH) : Galβ1-4GlcNAcβ1-3Galβ1-4GlcNAcβ1-3Galβ1-4Glc
iso lacto-N-neotetraose (iso LNnT) : Galβ1-4GlcNAcβ1-6Galβ1-4Glc
sialyl lacto-N-tetraose a (LST-a) : Neu5Acα2-3Galβ1-3GlcNAcβ1-3Galβ1-4Glc
sialyl lacto-N-tetraose b (LST-b) : Galβ1-3(Neu5Acα2-6)GlcNAcβ1-3Galβ1-4Glc
sialyl lacto-N-tetraose c (LST-c) : Neu5Acα2-6Galβ1-4GlcNAcβ1-3Galβ1-4Glc
disialyl lacto-N-tetraose (DSLNT) : Neu5Acα2-3Galβ1-3(Neu5Acα2-6)GlcNAcβ1-3Galβ1-4Glc
lacto-N-fucopentaose I (LNFP-I) : Fucα1-2Galβ1-3GlcNAcβ1-3Galβ1-4Glc
lacto-N-fucopentaose II (LNFP-II) : Galβ1-3(Fucα1-4)GlcNAcβ1-3Galβ1-4Glc
lacto-N-fucopentaose III (LNFP-III) : Galβ1-4(Fucα1-3)GlcNAcβ1-3Galβ1-4Glc
lacto-N-difucohexaose I (LNDFH-I) : Fucα1-2Galβ1-3(Fucα1-4)GlcNAcβ1-3Galβ1-4Glc
lacto-N-difucohexaose II (LNDFH-II) : Galβ1-3(Fucα1-4)GlcNAcβ1-3Galβ1-4(Fucα1-3)Glc
lacto-N-decaose (LND) : Galβ1-3GlcNAcβ1-3{Galβ1-3GlcNAcβ1-3[Galβ1-4GlcNAcβ1-6]Galβ1-4GlcNAcβ1-6}Galβ1-4Glc
fucosyl lacto-N-decaose I (F-LND-I) : Galβ1-3GlcNAcβ1-3{Galβ1-3[Fucα1-4]GlcNAcβ1-3[Galβ1-4GlcNAcβ1-6]Galβ1-4GlcNAcβ1-6}Galβ1-4Glc
lacto-diN-neotetraose (LdiNnT) : GalNAcβ1-4GlcNAcβ1-3Galβ1-4Glc
lacto-diN-neohexaose (LdiNnH) : GalNAcβ1-4GlcNAcβ1-3(GalNAcβ1-4GlcNAcβ1-6)Galβ1-4Glc
2'-fucosyllactose (2'FL) : Fucα1-2Galβ1-4Glc