氏名:山田 一作
1997年に東京都立大大学院工学研究科にて博士号取得。名古屋大学(日本学術振興会特別研究員(COE))などを経て、2002年から野口研究所研究員、2006年から同所にて糖鎖科学研究を開始し、糖鎖構造表記法、オントロジー、データベースなど糖鎖インフォマティクス研究に従事している。

氏名:安形 清彦
筑波大学大学院 生物科学研究科で学位取得後、La Jolla Cancer Research Foundation(現Sanford Burnham Prebys Medical Discovery Institute)にて、福田穰教授のもとで糖鎖生物学の研究について学ぶ。現所属(産総研)では、糖鎖遺伝子の発現解析、糖鎖の新しい機能の解析や糖鎖の応用について研究するとともに糖鎖DB(ACGG-DB)の開発に参画しています。

氏名:渡辺 由
2013年より新潟大学大学院医歯学総合研究科にて技術補佐員、研究員を経て、2018年より特任助手。主に、Web技術をベースにしたデータベースや解析ツールの開発に従事している。

氏名:小野 多美子
2017年9月より創価大学理工学部木下研究室の研究補佐員としてJSTの統合化推進プログラムに参加。GlyCosmosポータルの開発、データ収集に従事しています。
本シリーズの第6回では、複合糖質として糖脂質や糖タンパク質のデータベース、リポジトリについて説明いたします。多くのタンパク質は翻訳後修飾により糖タンパク質となり様々な機能を発現しています。そしてタンパク質を修飾する糖鎖は様々な構造が含まれています。また、疾患等により修飾される糖鎖構造が異なることも知られています。ここでは、複合糖質のデータベースおよび糖鎖・糖タンパク質の質量分析のデータリポジトリについて紹介します。
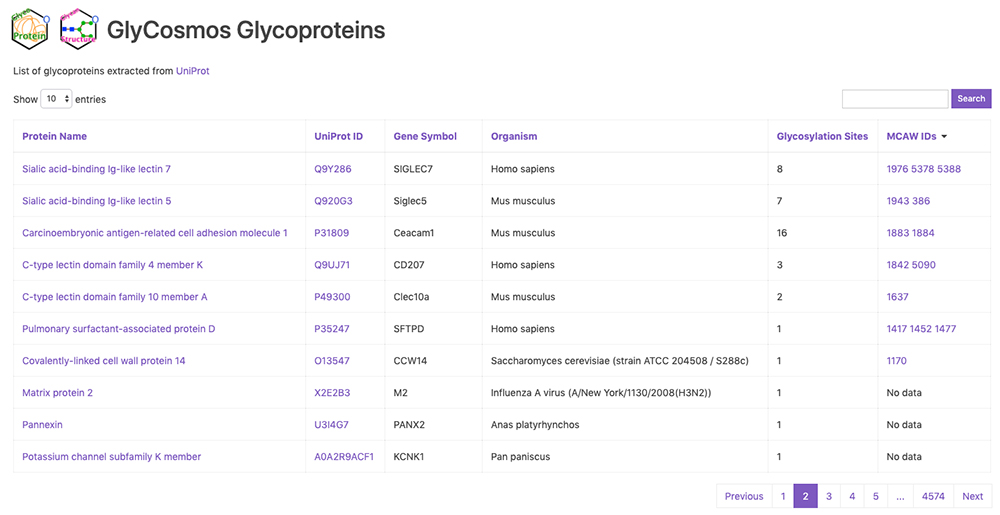
GlyCosmos GlycoproteinsはUniProt1から糖鎖修飾の注釈がつけられているタンパク質を抽出してまとめた糖タンパク質のリストです(図 1)。リストには、Protein Name、UniProt ID、Gene Symbol、Organism、Glycosylation siteの数が表示されています。また、UniProt IDで紐づけられたMCAW-DB2のMCAW IDsも表示されています。MCAW-DBについては、本シリーズレクチンのデータベースの現状で紹介されています。リスト右上のSearchからは、Protein Name、Gene Symbolのテキスト検索が可能です。
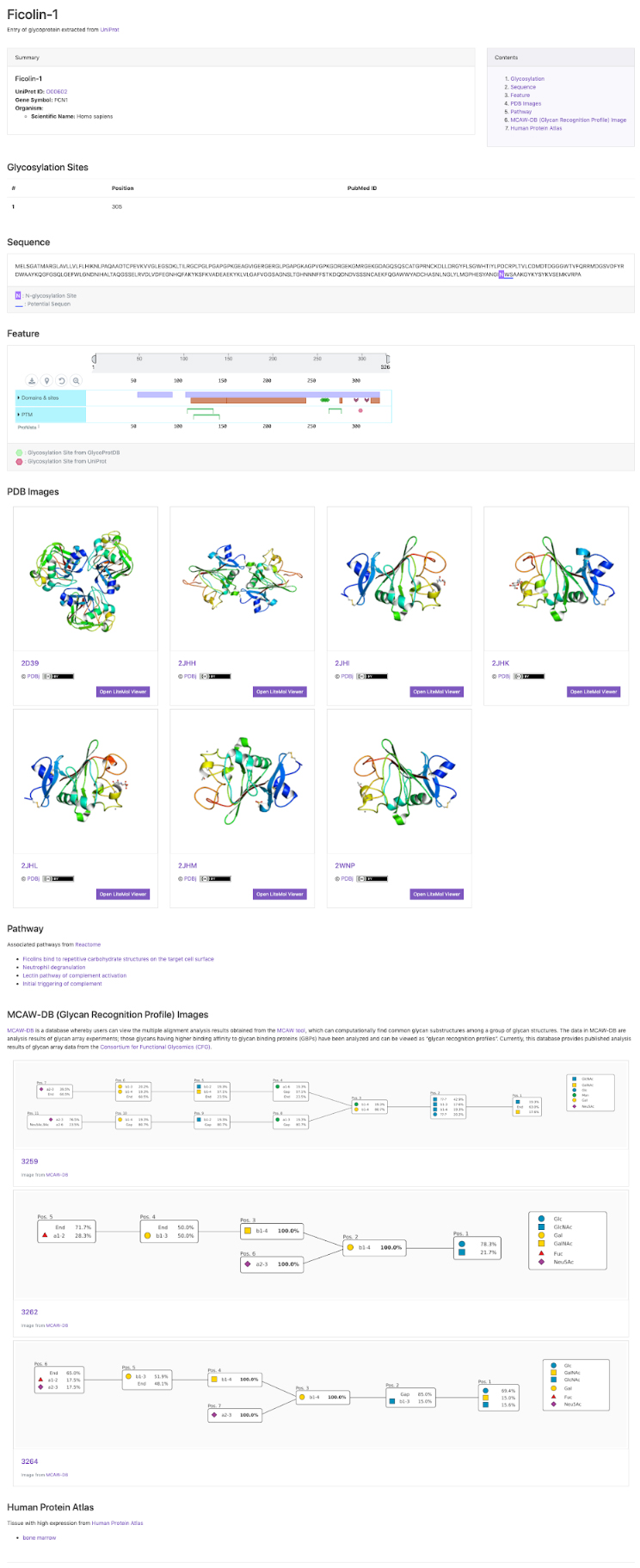
リストの Protein Nameをクリックすると、Glycoproteinのエントリーページ(図 2)に移動します。エントリーページには、Glycosylation Sites、Sequence、Feature、PDB Images、Pathway、MCAW-DB (Glycan Recognition Profile) Image、Human Protein Atlasといったコンテンツがあります(表 1)。
GlyCosmos GlycolipidsはLIPID MAPS Structure Database (LMSD)8から「"glyco", "glycan", "sugar", "saccharolipids"」のキーワードでピックアップしてまとめた糖脂質のリストです。GlyCosmos Glycolipidのトップページ(図 3)のLipid Classificationから検索したい糖脂質のカテゴリを選択します。カテゴリは階層になっており、最下層のカテゴリを選択すると糖脂質のリストが表示されます。リストには、Category、LM ID(LIPIDMAPS ID)、 Common Name, Systematic Name、Exact Mass、Formulaが表示されます。 LM IDをクリックするとLIPIDMAPSのページに飛びます。
| コンテンツ | 内容 |
| Glycosylation Sites | 糖鎖修飾部位と、文献情報がある場合はPubMed IDを表示しています。 |
| Sequence | 配列と共にN-glycosylation SiteとPotential Sequonの情報を表示しています。 |
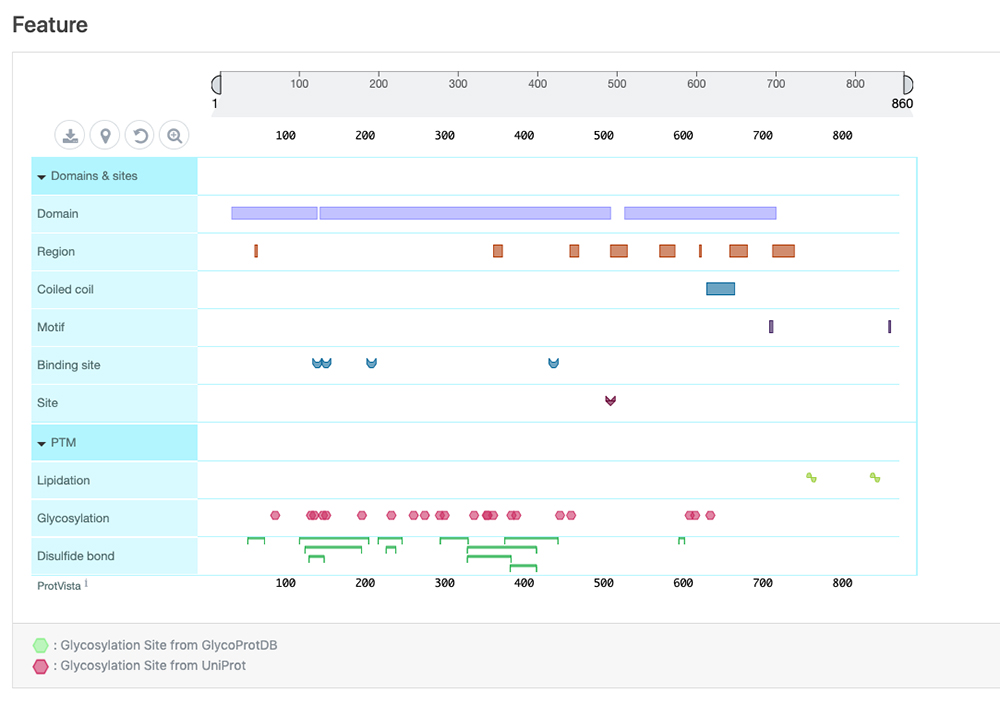
| Feature | タンパク質のドメインやアミノ酸修飾など、配列の注釈の情報を視覚的に確認することができます。この表示にはProtVista3というツールが用いられています(図 4)。 |
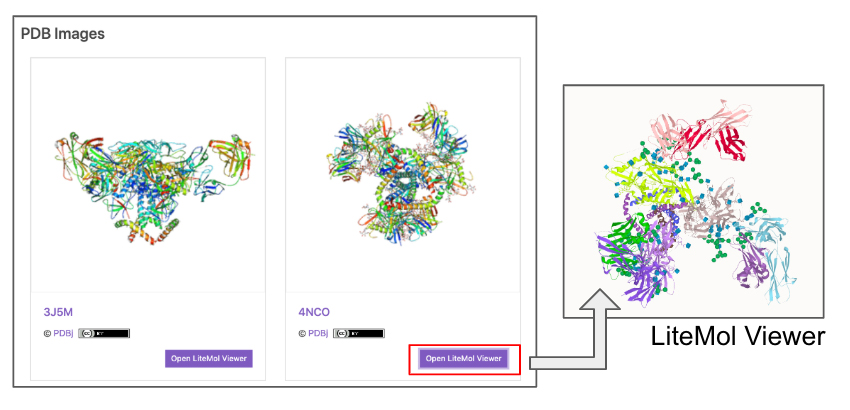
| PDB Images | Protein Data Bank(PDB)4の画像から立体構造について確認することができます。3Dで視覚化されているLiteMol Viewer5へのリンクがあり、より詳細に分子構造を確認することができます(図 5)。LiteMolでは糖鎖構造がSNFG形式で表示されています。 |
| Pathway | 糖タンパク質が反応に関与しているPathwayの一覧が表示されます。ここで表示させているPathwayはReactome6から抽出されています。クリックすると、GlyCosmos Pathwaysのページに移動してPathwayの詳細情報を確認することができます。 |
| MCAW-DB (Glycan Recognition Profile) Image | MCAW-DBのアラインメント結果が表示されています。 |
| Human Protein Atlas | 生物種がHomo Sapiensの場合、臓器別の細胞局在を確認することできます。HumanProteinAtlas7で発現量がHighとなっている臓器の一覧を表示させており、HumanProteinAtlasへのリンクがついています。 |

最近ではプロテオーム解析が進みデータが蓄積されたため、修飾を含めたプロテインデータベースが多数報告されています。しかしながら、糖鎖結合部位や糖鎖の構造を含めたデータベースは限られています。Unipep の様に同定されたN型糖鎖を含むペプチドのデータベースや、糖鎖修飾部位の情報を含むUniProtKBや糖鎖の組成を含むGlyConnectなどから情報を得ることが可能です。糖鎖構造に着目して質量分析法によって同定したN型糖鎖付加部位をデータベース化したのがGlycoprotein Database(GlycoProtDB, Kaji et al. 2012)で、日本糖鎖科学総合データベース(JCGGDB)の一つとして公開されました。現在では、表示方法などインターフェースや記載内容などを変更した最新版をACGG内で公開していますので、その特徴について紹介いたします。
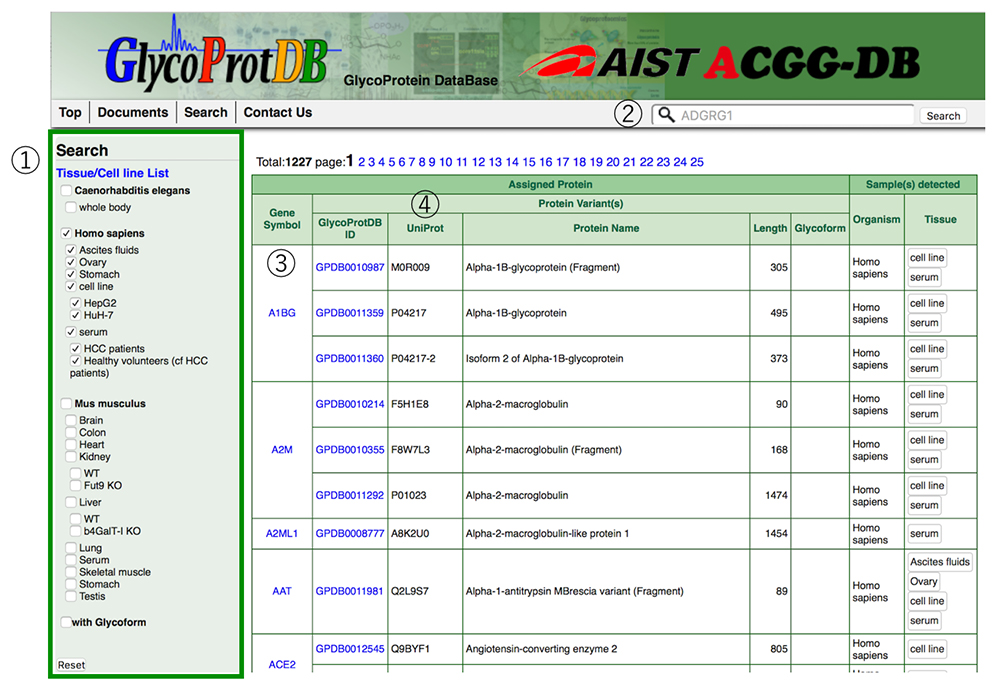
GlycoProtDBでは、線虫、ヒト、マウスの組織や細胞および血清などから取得したデータをリストしていますが、左側にあるカラムから取得したい組織や細胞を選択し、アルファベット順に表示することができます(図 6)。また、興味のある糖タンパク質を入力することでデータを検索することも可能です。
検索結果から糖タンパク質を選択すると、詳細ページにて糖タンパク質上のN型糖鎖付加部位やアミノ酸配列を見ることできます(図 7、8)。GlycoProtDBの主要な特徴として、組織やレクチン結合性による違いを見ることができます。
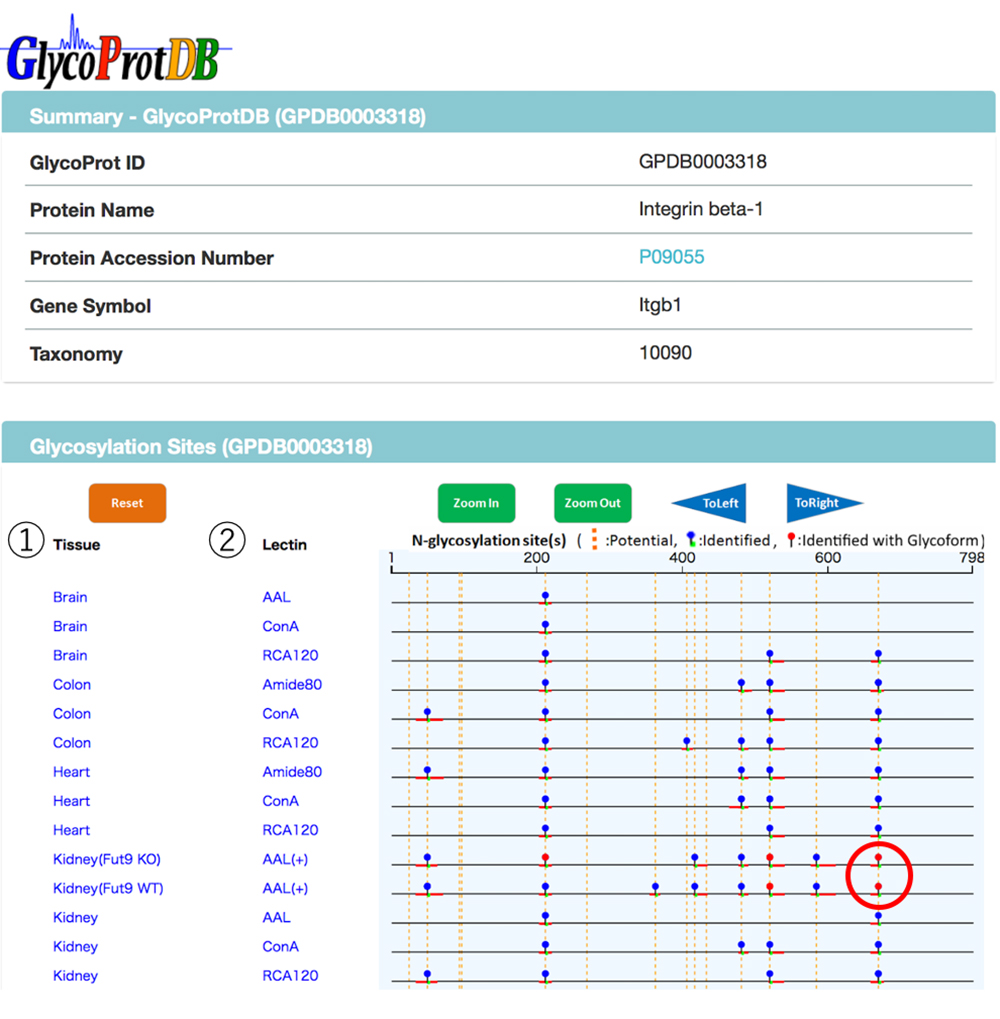
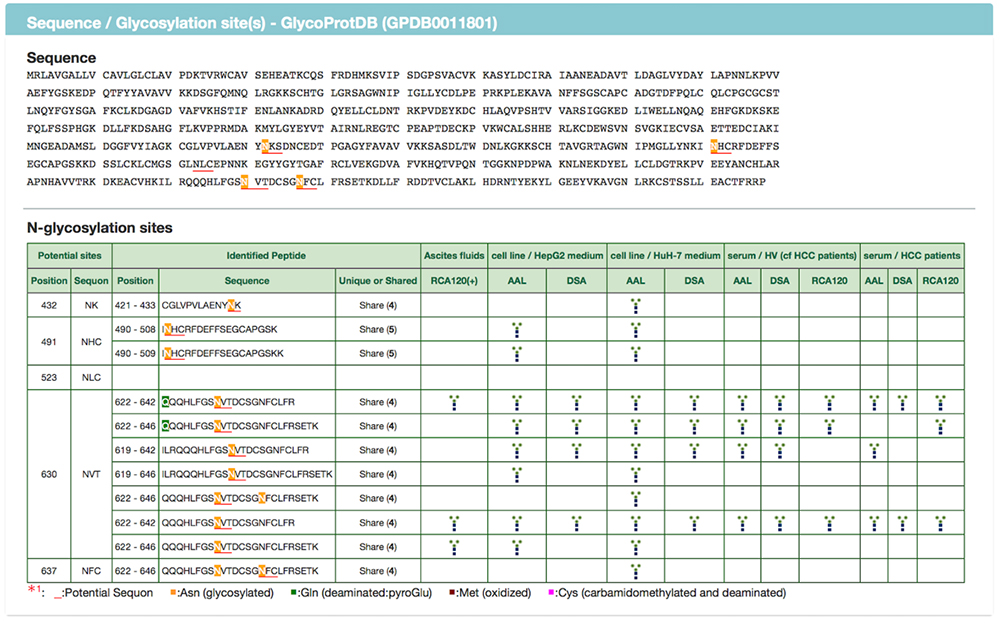
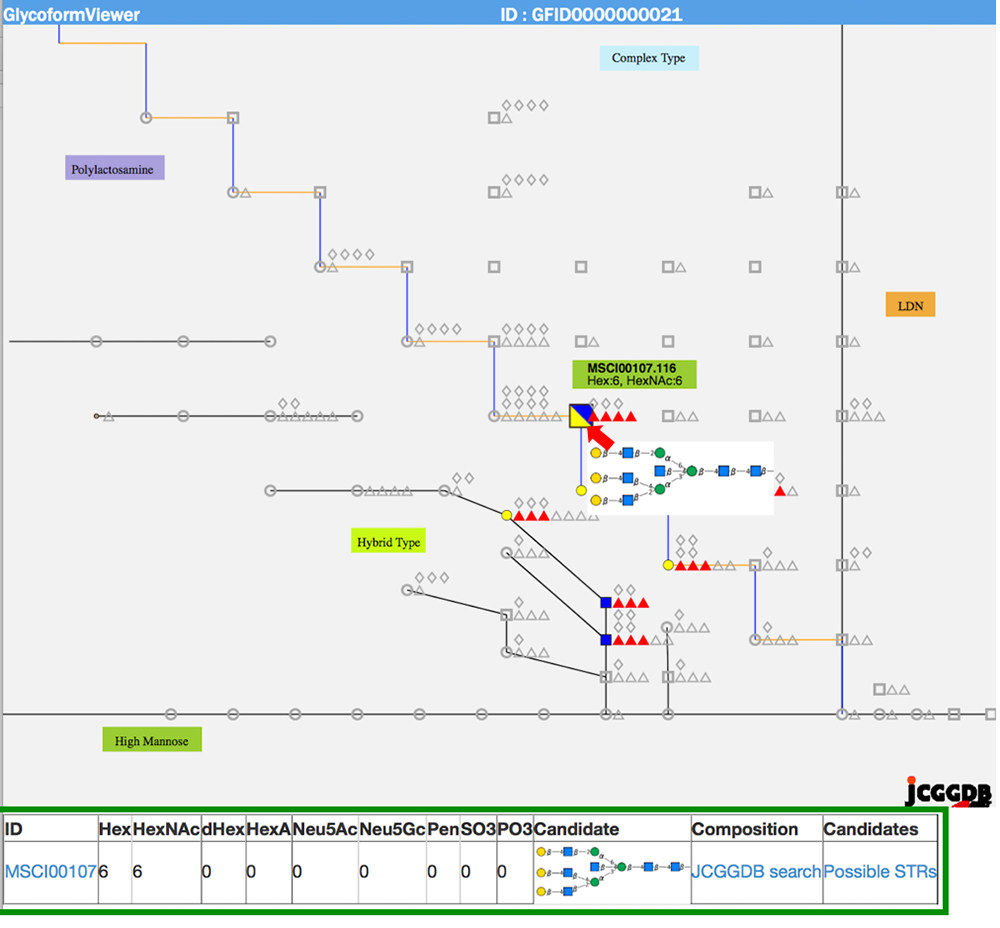
同じレクチンで認識される糖鎖構造を持つ部位をサンプル間で比較すると、組織によって差があることが分かります。また、糖鎖を糖タンパク質から切り離すことなく解析(GlycoRidge法)した結果は赤いピン(図 7)で表示されています。赤いピンを選択することで、どんな糖鎖構造(糖鎖組成)を持っているかをビューワーにて見ることが可能です(図 9)。ビューワー上では同定された糖鎖の上にマウスオーバーすることで候補の糖鎖構造を表示します。すなわち、GlycoProtDBではN型糖鎖の付加部位の実際を知るだけでなく、糖鎖構造の違いをも含めて組織、細胞や血清による差を容易に見ることができるデータベースです。
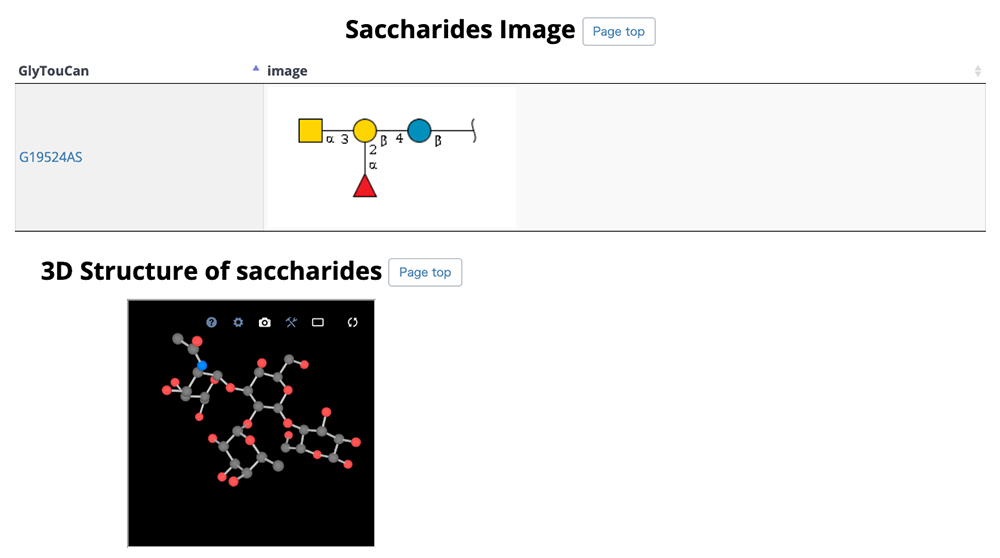
GlycoNAVIは糖鎖科学研究をサポートするためのウェブサイトです。ここでは生体高分子の立体構造データベースであるProtein Data Bank(PDB)11に含まれるデータを解析し糖鎖関連データを整理した二次データベース(TCarp)について紹介します。PDBには糖タンパク質、糖脂質、遊離糖鎖などの立体構造が収録されています。GlycoNAVIのトップページのGlycansサイトから糖鎖構造の一覧である図 10にアクセスすることができます。糖鎖構造は一覧表を順次閲覧するか、糖鎖構造リポジトリGlyTouCan12のアクセッション番号か糖鎖構造表記法であるWURCS13を入力することで検索することができます。GlyTouCanのアクセッション番号をクリックするとGlyTouCanのエントリーページが表示されます。

図 10で目的の糖鎖構造のWURCS文字列をクリックすると、その糖鎖構造を含むエントリーページのリスト図 11が表示されます。このリストでは立体構造、GlyTouCanのアクセッション番号、糖鎖構造のSNFG形式(https://www.ncbi.nlm.nih.gov/glycans/snfg.html)14が表示されます。先ほどと同様に左上のプルダウンで1ページの表示数の変更、右上のSearchで検索ができます。このリストにあるIDをクリックすると詳細ページが表示されます。

また、GlycoNAVIのトップページのProteinsサイトから糖鎖構造の一覧である図 12にアクセスすることができます。このリストには含まれる糖鎖の数、PDBのタイトルなどが表示されます。先ほどと同様に左上のプルダウンで1ページの表示数の変更、右上のSearchで検索ができます。このリストにあるIDをクリックすると詳細ページが表示されます。

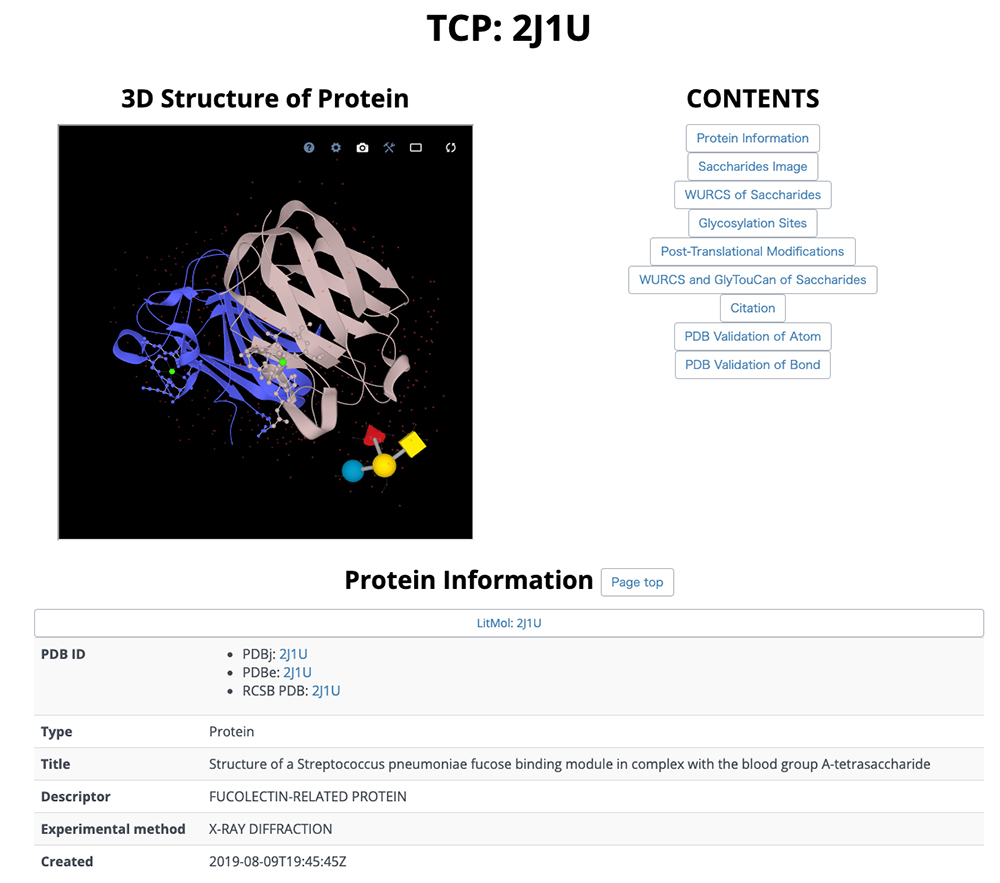
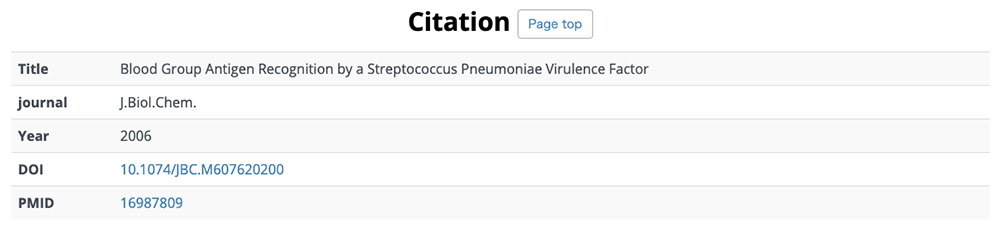
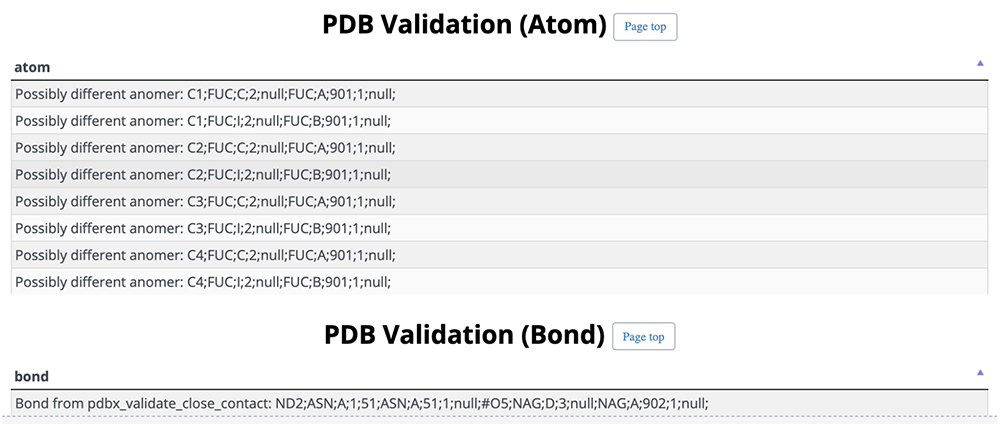
詳細ページでは、糖鎖構造を3D-SNFG15で描画した分子の三次元構造表示、PDBへのリンク、PDBエントリーのタイトル・説明、実験方法、解析日時(図 13)、GlyTouCanのアクセッション番号とリンク、糖鎖構造のSNFG表記、糖鎖の立体構造(図 14)、文献、PubMedへのリンクやデジタルオブジェクト識別子(DOI)とリンク(図 15)を得ることができます。また、図 16では、データ解析した際の糖鎖に関する検証結果を表示しています。この検証結果が表示されていると糖鎖の立体構造に注意すべき個所があることになります。
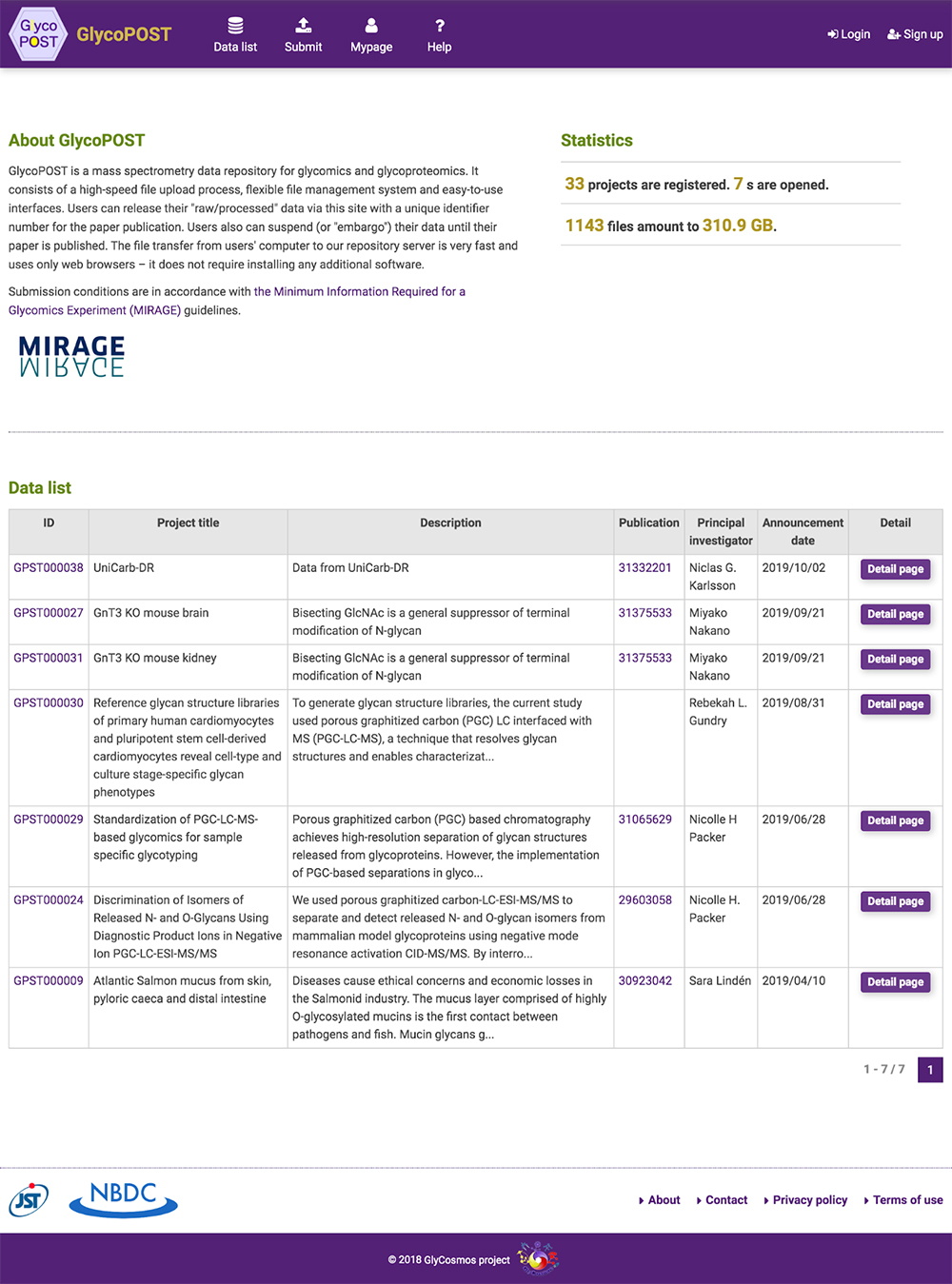
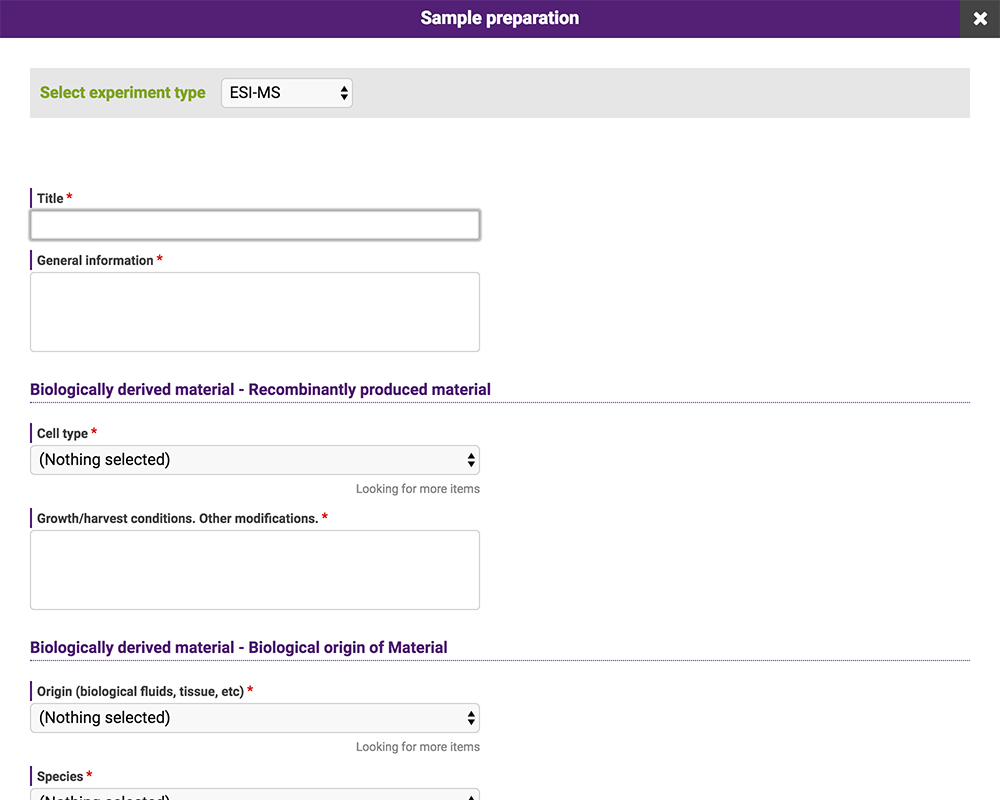
GlycoPOSTは、糖タンパク質の質量分析データを投稿するためのリポジトリデータベースです。Webブラウザからhttps://glycopost.glycosmos.org/にアクセスして利用します(図 17)。ユーザーは、自身の実験データを登録することも、他のユーザーが登録したデータを閲覧・ダウンロードすることもできます。多くの場合、論文発表に際して関連する実験データを公開する目的で利用されます。 リポジトリに投稿されるデータは、実験条件などのメタデータとファイルです。メタデータの入力は、プルダウンメニューから選択したり、テキストボックスに記述して行います(図 18)。入力項目は、MIRAGE Project16の提唱する糖鎖関連実験を報告する際のガイドラインに準拠しているため、国際的に有用とされる基準を満たしていることになります。ガイドラインに準拠する他のデータベース・リポジトリサイトとの互換性もあり、Excelファイルを介してインポートおよびエクスポートが可能です。
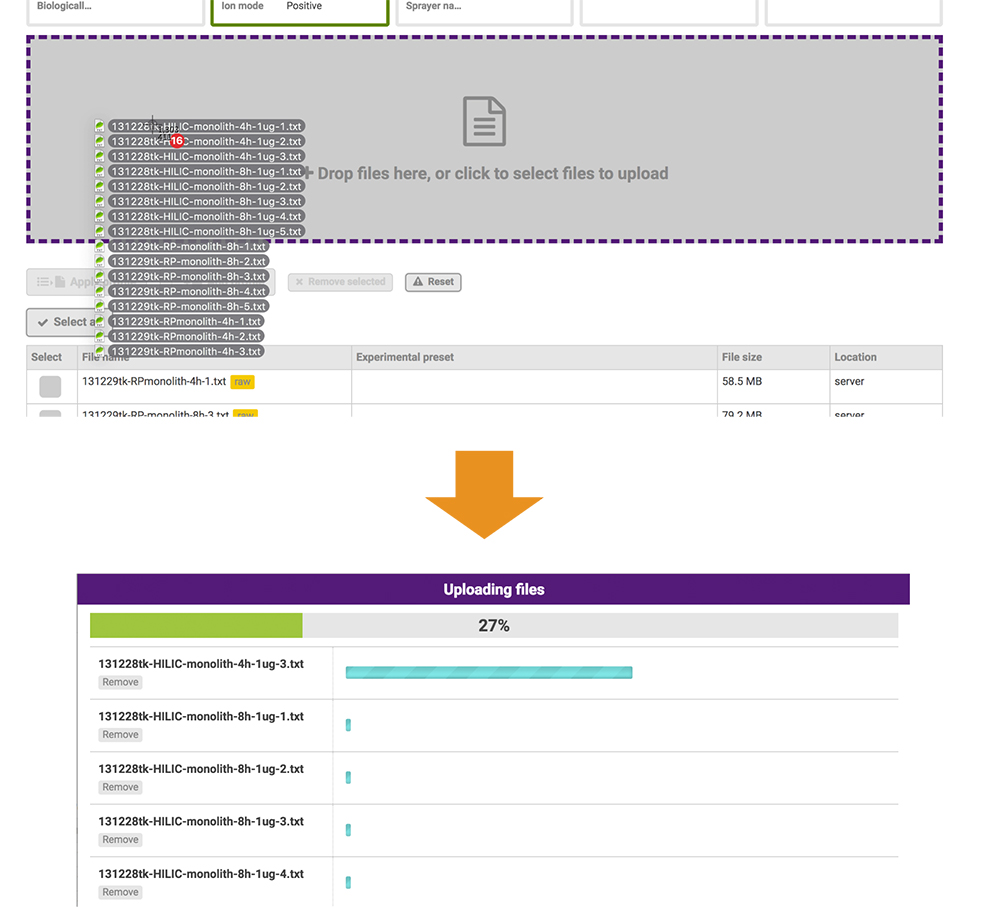
投稿されるファイルは、質量分析計から出力された生データやピークリスト、同定結果などです。GlycoPOSTのファイルアップロードシステムは、独自に開発したPRESTOというJavaScriptライブラリを使用しており、Webブラウザの標準機能を拡張することで、従来より高速なアップロードを実現しています(図 19)。この機能により、データ投稿のすべてのプロセスをウェブブラウザだけで実施できるようになっています。
具体的な投稿手順や用語についてはhttps://glycopost.glycosmos.org/helpに解説しています。