
氏名:木下 聖子
木下聖子は1999年に米国ノースウェスタン大学よりコンピュータ工学の博士号を取得。台湾のアカデミアシニカ(中央研究所)のポスドクを経て、ロサンゼルスにあるBioDiscovery社にて上級ソフトウェア開発者として3年間勤務。2006年からは京都大学化学研究所バイオインフォマティクスセンターに移り、糖鎖インフォマティクス研究を開始。現在は創価大学教授として、教育と研究を続けながら糖鎖コミュニティのために便利な糖鎖インフォマティクスツールを開発し、生体システムにおける糖鎖の機能解明へ応用している。
本稿は、糖質科学研究のためのバイオインフォマティクスリソースに関する一連のシリーズの最初を飾る論文である。本稿では、以降の論文を読む上で有用となる背景知識を簡単に紹介する。特に、糖鎖の表示に汎用されているテキスト及び記号命名法を、推奨する表示法とともに、記載する。次に、本シリーズで紹介するリソースの種類について簡潔に説明する。糖鎖関連ウェブリソースは、統合的データベースから、ウェブサービス、ウェブポータルまで多岐にわたる。このようなデータ網からの抽出作業は困難であり、本稿は、これらのリソースの現状に関する情報を、ユーザーに理解しやすく整理しようとするものである。糖タンパク質だけでなく糖脂質、プロテオグリカン、GPIアンカーなども網羅する「複合糖質」の項を設けているが、現行のデータベースは一般的に糖タンパク質のみを対象としていることに留意されたい。ただし、他の種類の複合糖質が忘れ去られているというわけではなく、今後の開発の課題となっている。
各種糖鎖の命名法についての論文レビューは多数あるが、本稿では、推奨する命名法について同様に述べる。
汎用性から、コミュニティが推奨する糖鎖構造テキストフォーマットは、主に、CarbBank、IUPAC、GlycoCT及びWURCSの4つである。CarbBankは同一名のデータベースに用いられているオリジナルフォーマットで、IUPACによって推奨されている。一方、IUPACは、CarbBankを含め、人に解読可能な3種類の糖鎖表示法を推奨している。GlycoCT及びWURCSは糖鎖インフォマティクス研究者が用いる主要なフォーマットである。ここでは、簡潔かつ人に解読可能な表示法としてIUPACの “condensed form”を用いることを提案する。ただし、人による解読性よりも正確性を重視する場合は、GlycoCT又はWURCSを推奨する。また、最近では、糖鎖構造の厳密な明記が必ずしも必要でない場合、GlyTouCan(後述)識別子(Tiemeyer et al. 2017b)を用いて、フォーマットと関係なく糖鎖を特定することができる。
糖鎖構造データベースのために考案された初期の糖鎖テキストフォーマットがCarbBankフォーマットで、これは糖鎖の二次元構造をタイプライターフォント(等幅フォント)テキストで示していた。図1にCarbBankフォーマットで表示した糖鎖構造の例を示す。このフォーマットは、人は読めるが、縦線を隣接単糖とすぐに結び付けられないため、コンピュータにとっては構文解析が困難である。
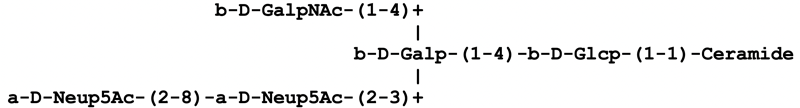
IUPAC(国際純正・応用化学連合)はhttps://www.qmul.ac.uk/sbcs/iupac/2carb/において、2-Carbと呼ばれる複合糖質の命名法を提唱している。特に「2-Carb-38. オリゴ糖構造定義のための記号の使用」の項では、オリゴ糖構造の表示法として、(1)単糖残基は3文字の記号で表す、(2)「還元基」すなわち、遊離ヘミアセタール基またはその修飾を有する残基(アルジトール、アルドン酸、グリコシドなど)は右端に置き、非糖質部分(タンパク質、ペプチドまたは脂質)へのグリコシル結合がある場合は、関与しているグリコシル残基を右に置くという2つの基本原則を推奨している。さらに、オリゴ糖に関しては3種類の糖質命名法が推奨されている。これらについて以下にまとめる。
拡張型(extended form):これは、CarbBankが採用しているフォーマットで、単糖単位の各記号はアノマー記述子及び立体配置記号によって表される。環サイズはフラノースがイタリックのf、ピラノースがイタリックのpなどと表される。結合しているロカントは糖記号間の括弧( )内に示され、両矢印は2つのアノマー位の結合を表す。例えば、α-D-Galp-(1→6)-α-L-Glcp-(1↔2)-β-D-Frufはラフィノースを表す。
凝縮型(condensed form):このフォーマットは、簡潔ながら人が解読できる糖質表示として、様々な文献、データベース及びウェブページにおいて最も汎用されている。このフォーマットでは、立体配置はD(通常はL型であるフコース及びイズロン酸を除く)、また特に指定のない限り、環はピラノース型と仮定して、立体配置記号及び環サイズを表す文字を省いている。さらに、アノマー記述子をロカントと共に括弧()内に記載する。ラフィノースを例に取ると、その構造はGal(α1-6)Glc(α1-2β)Frufと表示される。
短縮型(short form):このフォーマットは、さらに、アノマー炭素原子のロカント、結合ロカントを囲む括弧( )及びハイフンも省いたものである。分岐は括弧( )または角括弧[ ]を用いて同一線上に示される。必要な場合は、より具体的な表示のため、立体配置記号及び環サイズ指定子などを入れることもできる。以上より、ラフィノースは短縮形ではGalα-6Glcα-βFrufまたはGalα6GlcαβFrufとなる。
GlycoCTは、ドイツがん研究センターのClaus Wilhelm von der Lieth博士が初めに率いたEuroCarbプロジェクトの下で開発されたフォーマットである(Ranzinger et al. 2009)。本フォーマットは、最初の糖鎖構造統合データベースの1つであるGlycomeDB (Herget et al. 2008)で用いられているフォーマットであるため、現在、バイオインフォマティクス研究者の間で最も広く用いられている。GlycomeDBのデータ管理が困難なため、現在では国際糖鎖構造リポジトリであるGlyTouCan (Tiemeyer et al. 2017a)に統合されている。図 1の構造はGlycoCTでは図 2のように表示されている。

一方、GlyTouCanの開発初期になされた、利用すべき糖鎖表示フォーマットに関する討論で、GlycoCTに関するいくつかの問題が提起された。GlyTouCanはSemantic Web技術を用いて開発されているため、糖鎖表示に関する最初の要件の1つは、糖鎖が直鎖で表すことであった。次の要件は、ライブラリを使用せずにあいまい構造を表すことが可能なことであった。例えば、単糖上の新たな置換は、既知か否かにかかわらず表示されなければならない。このように、修飾を名称ではなく化学物質で特定しなければならなかった。当時は、既存の糖鎖表示法で、これらの要件を満たすものはなかったため、WURCS(Web3.0 Unique Representation of Carbohydrate Structures)が開発された(Matsubara et al. 2017)。WURCSは、全ての糖鎖表示が特有で、固有の識別子で表示されるための規則を定めている。図 3はWURCS表示法の1例である。GlycoCTの残基は角括弧[ ]で示され、その後スラッシュ/が続き、連結と立体配置を示している。テキスト「NCC/3=0」を含む残基はNアセチル化修飾を表している。
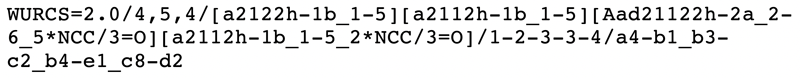
糖鎖の構造は複雑なため、多くの糖鎖生物学者はその表示に図を用いている。CarbBank表示に加え、現在用いられている記号での主な糖鎖表示法には、Symbol Nomenclature for Glycans(SNFG)とオックスフォード表示法の2つの方法がある。SNFGは最近、コミュニティから多くの支持を受けたが、それは主に、単糖表示の記号に関する推奨である。一方、オックスフォード表示法では、アノマー型と炭素位を区別するためのグリコシド結合表示法についても規定している。このように、SNFG表示法は単糖の記号について推奨しているが、グリコシド結合の表示法はユーザーに任せられている。
SNFGの記号群は、国際的に著名な糖鎖生物学者、化学者及び情報学者から成るワーキンググループの討議に基づいて考案された。現在推奨されている記号の一覧はhttps://www.ncbi.nlm.nih.gov/glycans/snfg.htmlで閲覧可能である。これは元々「CFG命名法」と称されることの多い「Essentials of Glycobiology (Varki et al. 2017)」で推奨された記号に基づくものである。
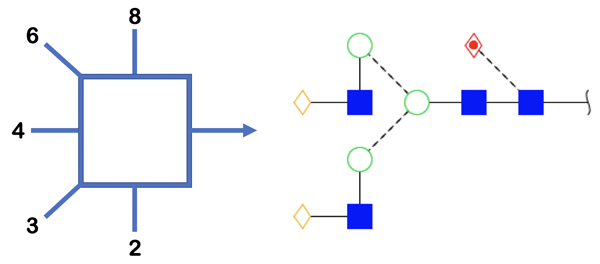
オックスフォード法は、2009年にオックスフォード大学糖鎖生物学研究所の研究者が考案したもので、単色のみで単糖を明確に区別できるような記号を用いている。グリコシド結合の表示ではα立体化学が点線で、β立体化学が実線で表される。また、結合の表示では、結合部位の炭素位を表すために角度も用いている。図 4に、グリコシド結合配座を表す様々な角度を示す。
本シリーズでは、様々な糖鎖関連ウェブリソースについて、特に、GlyCosmosポータルサイトからアクセスできるものを紹介していく。ユーザーが、関心のあるウェブリソースを最も効率的に見つけることができるように、GlyCosmos同様、生物学的コンテンツに基づいて各データベースをカテゴライズした。本稿では各ウェブリソースについて簡単に紹介し、本シリーズを通して各リソースの利用法について紹介していく。ただし、データベースとリポジトリの違いに留意されたい。データベースは主に、ユーザーによる変更が不能な、精選されたコンテンツから成るが、リポジトリは、固有のアクセッション番号を得るために、ユーザーがデータを追加することができる。本シリーズでもこのように区別して用いる。
糖鎖と最も緊密な関連のある遺伝子は、糖鎖を合成する糖転移酵素や糖質加水分解酵素などの糖酵素をコードする遺伝子及び糖ヌクレオチド輸送体など糖鎖の生合成を助ける遺伝子である。本シリーズでは、ACGG-DBの提供するGlycoGene Database(GGDB)について述べる。ただし、フランスのCarbohydrate Active Enzymes(CAZy)データベース(Terrapon et al. 2017)も極めて有名で、これらのデータを他のデータベースと効率的に統合するため、CAZyとGlyCosmosの連携が進行中である。
糖鎖関連タンパク質は、糖鎖とどのように相互作用するかによって、(a)糖鎖遺伝子のタンパク質産物、(b)糖鎖が結合して糖タンパク質やプロテオグリカンとなるコアタンパク質及び(c)細胞表面上の糖鎖を認識・結合する糖鎖結合タンパク質にカテゴライズされる。(a)のタンパク質は通常Glycogenesに含まれている。(b)を含むデータベースには、ACGG-DBのGlycoProtDB 、UniCarbKB及びGlyConnectなどがある。一方、より包括的なタンパク質データベースであるUniProtKB及びPDBにも、糖修飾部位などのグリコシル化データが含まれている。(c)はACGG-DBのLectin frontier Database(LfDB)に含まれているが、糖鎖アレイ実験のデータは欧米の各種データベースにも含まれており、これらは現在、それぞれのプロジェクトで統合が進んでいる。GlyCosmos Portalでは、これらのデータは複合糖質のカテゴリーに含まれている。
糖鎖関連疾患は(a)糖鎖遺伝子がコードする糖鎖関連タンパク質の変異または(b)糖鎖及び糖タンパク質と相互作用する病原体によって生じる。これらの情報を含むデータベースには、いずれもACGG-DBが提供するGlyco-Disease Gene Database(GDGDB)及びPacDBがある。
糖鎖データベースは元々、糖鎖構造に経路、疾患などのアノテーションを追加するために開発された。国際糖鎖構造リポジトリであるGlyTouCanは現在、GlyTouCan IDをアクセッション番号として用いることにより、全ての糖鎖データをリンクさせることができる中心的なリソースとして用いられている。GlyCosmosでは、いずれも質量分析法で得られたグライコミクスデータを視覚化するTotal Glycome Database及びGlycomeAtlasへのアクセスも可能である。複合糖質とは糖タンパク質及び糖脂質のことであり、糖タンパク質とは糖鎖が結合したタンパク質であり、糖脂質とは糖鎖が結合した脂質である。いずれも細胞表面に存在することが多い。GlyCombは、現在、糖鎖用のGlyTouCanと同様、複合糖質用のリポジトリとして開発中で、今後、グライコミクス、プロテオミクス及びリピドミクス間を結ぶ重要なリンクとなるだろう。そのための第一段階として、グライコミクス及びグライコプロテオミクスデータの質量分析リポジトリとして、GlycoPOSTが現在利用可能である。
糖鎖関連データリポジトリに関しては、現在、2つのリポジトリが公開されており、本稿執筆時点でさらに1つのリポジトリが開発中である。GlyTouCanは糖鎖構造リポジトリで、単糖組成、糖鎖フラグメント、曖昧なリンケージまたは単糖など、全ての糖鎖エントリに固有のアクセッション番号を付与する。何らかの形態の糖鎖に関する原稿の発表準備をしているユーザーは、それを登録し、固有のアクセッション番号を得ることができる。現在利用できる2つ目のリポジトリは、グライコミクス及びグライコプロテオミクスの質量分析(MS)生データのリポジトリであるGlycoPOSTである。UniCarb-DRとMIRAGE(次項参照)のコラボレーションで、GlycoPOSTはMSデータに固有のアクセッション番号を付与し、MIRAGEが推奨するように、メタデータとともに保存する。
前述したように、MIRAGE(minimum information required for a glycomics experiment)は、糖鎖関連実験データの報告に関するガイドラインを提供するイニシアチブである。本稿執筆時点で、MIRAGEガイドラインは質量分析(Kolarich et al. 2013), 糖鎖アレイ(Liu et al. 2017)、試料調製(Struwe et al. 2016)及び液体クロマトグラフィ(準備中)を対象としている。
インフォマティクスに関しては、現在、グライコミクスデータの解析に多数のソフトウェア及びツールが利用されているが、多くは、GitHubなどのオープンソースリポジトリで公開されている。しかし、これによって、糖鎖インフォマティクス研究者は他のリソースを見つけることが困難になっている。Glycoinformatics Consortium(GLIC)は、コミュニティが関連のあるソフトウェアやデータベースを見つけられるよう、ソフトウェア、データベース及びディベロッパー情報のセントラルリポジトリを提供する目的で設立された。GLICは、科学者がソフトウェアツールの依頼を提出するためのフォームも提供している。
GlyGenは、米国国立衛生研究所コモンファンドプログラムが出資するプロジェクトで、各種オミックスをグライコミクスと統合するプロジェクトである。GlyGenは、UniProtに登録された(糖)タンパク質に基づき、ヒト及びマウスについては公表されているように、糖タンパク質、糖鎖及び各種プロテオフォームを提示している。
ExPASy(Swiss Institute for Bioinformatics’ Bioinformatics Resource Portal)は、プロテオミクス、ゲノミクス、系統学、システム生物学、集団遺伝学、トランスクプトミクスなど、ライフサイエンスの各分野において科学データベース及びソフトウェアツール(すなわち、リソース)へのアクセスを提供する。Glycomicsのページを設け、国内外で開発されたソフトウェア及びデータベースの一覧を提供し、グライコミクス研究を支援している。これらの最新データベースの1つがGlyConnectである。これは、糖タンパク質、部位特異的グリコシル化パターン及びそれらを修飾する糖鎖を視覚化することにより、グライコミクスとプロテオミクスを統合することを目指している(Alocci et al. 2019)。
GlyCosmosは、科学技術振興機構及びバイオサイエンスデータベースセンターが出資する統合プロジェクトである。Semantic Webテクノロジーを用いて、糖鎖、それらの遺伝子、タンパク質並びに関連経路及び疾患が、ユーザーフレンドリーな方法で統合・視覚化されている。
これら3つのプロジェクト(GlyGen、Glycomics@ExPASy及びGlyCosmos)は、2018年夏にGlySpace Allianceとして、お互いのデータを共有することに同意した。これらは類似したプロジェクトで、重複している可能性があるため、ユーザーが混乱し、分散する懸念があった。全てのデータをまとめて提供するため、GlySpace Allianceのメンバーは、お互いにデータを共有するためにオープンソースライセンス及びソフトウェアを用いることに同意した。最新かつ正確なデータを提供するため、年次総会を開き、統合可能な各種データに関して議論される。