
氏名:五十嵐 学
北海道大学人獣共通感染症リサーチセンター
1997年北海道大学工学部合成化学工学科卒業。2000年北海道大学大学院工学研究科分子化学専攻修士課程修了。2004年北海道大学大学院医学研究科生体機能学専攻博士課程修了。2001年4月より日本学術振興会特別研究員(DC1)。2004年4月より北海道大学大学院医学研究科COE研究員(プログラム名「人獣共通感染症制圧のための研究開発」)。2005年12月より北海道大学人獣共通感染症リサーチセンター国際疫学部門博士研究員。

氏名:伊藤 公人
北海道大学人獣共通感染症リサーチセンター
1992年北海道大学工学部電気工学科卒業。1994年北海道大学大学院工学研究科電気工学専攻修士課程修了。1999年北海道大学大学院工学研究科電気工学専攻博士課程修了。1999年7月より北海道大学大学院工学研究科講師(中核的研究機関研究員)。2002年4月より北海道大学大学院工学研究科リサーチアソシエイト。2004年4月より北海道大学大学院情報科学研究科助手。2005年5月より北海道大学人獣共通感染症リサーチセンター国際疫学部門助教授(2007年4月より准教授)。

氏名:高田 礼人
北海道大学人獣共通感染症リサーチセンター
1993年北海道大学獣医学部獣医学科卒業。1996年北海道大学大学院獣医学研究科博士課程修了。1996年4月より日本学術振興会特別研究員(PD)。1997年4月より北海道大学大学院獣医学研究科助手。2000年10月より東京大学医科学研究所助手。2005年5月より北海道大学人獣共通感染症リサーチセンター国際疫学部門教授。
エンベロープウイルスの粒子表面には糖蛋白質が存在する。ヘンドラウイルス、SARSコロナウイルス、インフルエンザウイルス、HIV、肝炎ウイルス、ウエストナイルウイルスなどのウイルスでは、糖蛋白質に付加されている糖鎖がその生活環において非常に重要な機能(宿主細胞への侵入、蛋白質分解過程、蛋白質輸送など)を担っている1。また、ウイルス糖蛋白質の抗原性は糖鎖獲得変異や糖鎖欠失変異によって大きく変化する。インフルエンザウイルスやHIVでは、そのような変異が宿主中和抗体からの回避に影響していると考えられている1。この章では、インフルエンザウイルスの表面糖蛋白質であるヘマグルチニン(HA)に焦点をあて、抗原変異に伴って起こるN型糖鎖付加について述べる。
A型インフルエンザウイルスは、鳥類や哺乳類(ヒトを含む)に広く感染する人獣共通感染症病原体であり、その自然宿主はカモ目およびチドリ目に属する野性の水禽類であると考えられている。インフルエンザウイルスは粒子表面に2種類の糖蛋白質HAとノイラミニダーゼ(NA)を持ち、抗原性の違いからHAは16種類(H1-H16)、NAは9種類(N1-N9)の亜型に分類される2,3。野鳥で保持されているインフルエンザウイルスが、種の壁を越え、新たな宿主動物内(例えば、ブタ、ウマ、ニワトリ、ヒト)で十分に増殖する能力が備わると病原体として感染症を引き起こす。自然宿主以外の動物で流行が始まるとHAの抗原変異が急速に起こり、これに伴い新たな糖鎖が付加されることがある。
前世紀、人類は3度のインフルエンザ・パンデミック(世界的流行)を経験した(1918年H1N1:スペインかぜ、1957年H2N2:アジアかぜ、1968年H3N2:香港かぜ)4,5。パンデミックウイルスはその後、抗原性を少しずつ変化させながら、季節性インフルエンザとして毎年ヒトの間で流行を繰り返す。インフルエンザウイルスの抗原変異は主にHA蛋白質で起こる。HAはウイルス表面のスパイク状の蛋白質で、立体構造上、球状頭部と茎部に分けられる6(図1)。このうち球状頭部が中和抗体の主要な標的である。HA分子上のエピトープ(中和抗体が結合する領域)にアミノ酸変異が蓄積すると、抗体による中和作用を逃れたエスケープ変異株が出現する。これが抗原変異のメカニズムである。また抗原変異に伴い、HA上に新たな糖鎖が付加することがある。抗原領域に新たな糖鎖が付加すると、中和エピトープが覆い隠されることがあるので、糖鎖獲得は効率の良い抗原変異であると考えられている。
図 1 HAモノマーの3次元構造
本研究ではすべてのHA亜型においてアミノ酸残基番号52─277(H3 numbering)を球状頭部領域と定義した。
我々は過去にヒトの間で流行した3つの亜型のインフルエンザウイルス(H1N1、H2N2、H3N2)のHA遺伝子を大規模に解析し、N型糖鎖付加のコンセンサス配列(sequon: Asn-Xaa-Ser/Thr, XaaはPro以外)の数の変遷を追った(図2)。H3N2ウイルスは1968年に出現以来、現在まで約40年間ヒトで流行を続けている。HA球状頭部のsequonの数はこの間に増えていた7-9。すなわち、1968年のパンデミック時に分離されたプロトタイプ株(A/Hong Kong/1/68)のHA球状頭部にはsequonが2つしかなかったが、最近流行しているH3N2ウイルスには6〜7つのsequonがある(図2A)。H1N1ウイルスは1918年のパンデミック後、約40年にわたってヒトで流行を繰り返した。さらに、1977年に再出現してから、現在まで約30年間流行を続けている。最近流行しているH1N1ウイルスは、1918年のパンデミック時の感染者から検出されたプロトタイプ株(A/South Carolina/1/18)10より、HA球状頭部のsequonの数は多い9(図2B)。一方、H2N2ウイルスは、1957年に出現し、約11年間でヒトでの流行を終えた。H2N2ウイルスは、H3N2やH1N1ウイルスと異なり、HA球状頭部のsequonの数は変化していなかった(図2C)。
図 2 ヒトインフルエンザウイルスHAのsequonの数の変化
ヒトインフルエンザウイルス(A) H3N2、(B) H1N1、(C)H2N2のsequonの数を調べた。箱ひげ図の箱の中央の線は中央値を、箱部分は25%点と75%点を、箱の上下の先端は許容範囲(箱の長さの1.5倍)以内にある最大値と最小値を示している。○印は、はずれ値に相当する値である。解析にはそれぞれ3415、950、88本の塩基配列データを用いた。データはすべてNCBI Influenza Virus Resourceからダウンロードした。
上に述べた観察結果から、過去にパンデミックを起こしたインフルエンザウイルスがヒトの間で持続的に流行するには、抗原変異に伴いHAへ新たな糖鎖を獲得することが重要だったという仮説が浮かび上がってくる7,8,11。そこで、我々は、将来いずれかのHA亜型の鳥インフルエンザウイルスがヒトの新型インフルエンザウイルスとして出現すると仮定し、全てのHA亜型の鳥インフルエンザウイルスについて、HA遺伝子の塩基配列上に潜在する糖鎖付加ポテンシャルを解析した。
我々は、塩基配列上1〜3塩基置換でsequonとなるコドン(図3)の数を糖鎖付加ポテンシャルとして定義した12。図3に示すように、1塩基、2塩基および3塩基変異でsequonとなる9塩基のモチーフ配列をそれぞれCand1、Cand2、およびCand3と呼ぶ。
図 3 糖鎖付加ポテンシャルの定義
(A)Cand1(1塩基置換でsequonになる)のコドンの例。(B)、(C)はそれぞれCand2、Cand3の例。
我々は、自然宿主であるカモ目およびチドリ目に属する野性の水禽類から分離されたインフルエンザウイルス株について、全16HA亜型のHA遺伝子(球状頭部領域)中のsequon、Cand1、Cand2の数を調べ、その数を比較した。sequonの数はHA亜型間で相違は認められなかった(図4A)が、Cand1およびCand2の数は亜型によって有意に異なっていた(図4B、4C)。この結果は、自然宿主で保持されているインフルエンザウイルスが、宿主の壁を越え、新型インフルエンザウイルスとしてヒトの間で流行した場合、新たな糖鎖を獲得する能力はHA亜型により大きく異なる可能性を示唆している。
図4B に示すように、H2亜型のHA遺伝子は、H1やH3亜型と比べてCand1およびCand2の数が少なく、糖鎖付加のポテンシャルは低かった。これはパンデミック時のプロトタイプ株、A/South Carolina/1/18(H1N1)、A/Japan/305/57 (H2N2)、および A/Hong Kong/1/68 (H3N2)で比較しても同様の傾向を示した。上述のように、ヒトH3N2およびH1N1インフルエンザウイルスのHA球状頭部の糖鎖の数は、流行に伴い増えていた(図2A、2B)。一方、H2N2ウイルスでは、流行期間に、新たな糖鎖を獲得していなかった(図2C)。H2N2ウイルスが11年間で、ヒトの間での流行を終えてしまった理由は複数考えられているが、我々の解析結果ではH2亜型のHA遺伝子は糖鎖を獲得するポテンシャルがH3およびH1亜型のHAと比べて低かったことから、1957年にH2N2ウイルスがパンデミックを起こした時点ですでにヒトの間で長期間流行するための能力が低かった可能性が示された。
図 4 鳥インフルエンザウイルスHAのsequonの数と糖鎖付加ポテンシャルの比較
全16HA亜型の鳥インフルエンザウイルスの(A) sequon、(B) Cand1、(C)Cand2の数の比較。箱ひげ図については図2を参照。統計学的データは以前に述べた12。解析には、47 (H1)、70 (H2)、104 (H3)、113 (H4)、747 (H5)、177 (H6)、66 (H7)、10 (H8)、89 (H9)、25 (H10)、52 (H11)、21 (H12)、15 (H13)、2 (H14)、5 (H15)、および6 (H16)本の塩基配列データを用いた。
近年、H5、H7、H9亜型のインフルエンザウイルスが家禽の間で流行を繰り返している。1997年に香港でH5N1ウイルスのヒトへの感染例がはじめて報告されて以来、H5N1、H7N7、H7N3、H9N2亜型など家禽で流行しているインフルエンザウイルスが鳥からヒトへ直接伝播した例が多数報告されている4,13-15。これらの鳥インフルエンザウイルスがヒトの新型ウイルスとして出現することが危惧されている。我々の解析結果では、H5およびH9亜型のHA遺伝子にはH1やH3亜型と同程度の数のCand1があった。さらにCand2の数を比較すると、H5亜型はH1亜型よりも多くH3亜型と同程度であり、H9亜型はH1亜型と同程度であった。したがって、もしH5やH9亜型のウイルスが将来ヒトの間で流行をはじめたら、過去のパンデミックウイルスよりも糖鎖を獲得する能力は高いかもしれない。実際に、近年流行しているH5N1ウイルスのほとんどは家禽類で流行するうちに、158番目(H3 numbering)に糖鎖を獲得したことが報告されている16。一方、H7亜型のCand1およびCand2の数は、ともにH2亜型よりも少なかったことから、H7亜型のウイルスがヒトの間で流行をはじめた場合、糖鎖を獲得しつつ抗原変異する能力は低いかもしれない。
次に我々はH1、H2、H3、H5、H7、H9亜型について、Cand1のHA立体構造上の位置を調べた。遺伝子配列解析から得られた結果と同様に、H2亜型のHA球状頭部では、分子表面に存在するCand1の数はH3、H5、H9亜型よりも少なかった。また、鳥のH5およびH9亜型では、過去にパンデミックを起こしたH1N1、H2N2、H3N2ウイルスのプロトタイプ株よりも表面に存在するCand1の数は多く、H7亜型ではその数はもっとも少なかった。これらの結果よりHA分子表面に露出しているCand1の数も亜型によって異なることを確認した。
図 5 HAの3次元構造上のsequonとCand1の位置
HAの3次元モデル。(A) H1 (A/South Carolina/1/18)、(C) H3 (STRAIN X-31)、(D) H5 (A/duck/Singapore/3/97)、(E) H7 (A/turkey/Italy/02)の構造はProtein Data Bankから得た。PDB codeはそれぞれ1RUZ、1HGF、1JSM、1TI8。(B) H2 (A/Japan/305/57)、(F) H9 (A/duck/Hokkaido/9/99)の構造はホモロジーモデリングにより作成した。青はsequonのAsn残基を表している。赤はCand1の中で、1塩基変異でsequonになる残基を示している。残基番号はH3 numberingである18。括弧の中の番号は、sequonとなったときのAsn残基番号を示している。
最後に我々は、H3N2やH1N1ウイルスがヒトで流行する間に獲得したsequonが、それぞれのプロトタイプ株のHA遺伝子配列上でCand1、Cand2、Cand3のいずれに由来していたかを調べた。
現在のH3N2ウイルスがヒトでの流行中に獲得したsequonはすべて、1968年に出現した時点のプロトタイプ株A/Hong Kong/1/68のHA遺伝子配列上ではCand1、Cand2、Cand3のいずれかであった(図6)。最初の20年間で獲得したsequon(Asn残基番号:63、126、246)は、1968年の株の配列上で、すべてCand1だった。1997年に獲得した2つのsequon(Asn残基番号:122、133)はともにCand2、2003年に獲得したsequon(Asn残基番号:144)はCand3由来であった。
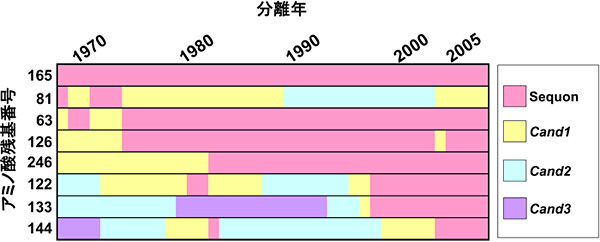
図 6 ヒトH3N2インフルエンザウイルスHAの糖鎖付加部位のコドンの変遷
データは各年代で多数をしめた結果である。
H1N1ウイルスでも同様にコドンの変化を調べた(図7)。ヒトH1N1ウイルスのプロトタイプ株、A/South Carolina/1/18には、HA球状頭部に1つだけsequon(Asn残基番号:94A)が存在していた。また1977年にヒトの間に再出現したH1N1ウイルスは94A、131、158、163、271番目の5つのAsn残基を含む配列がsequonとなっていた。さらに、最近流行しているH1N1ウイルスでは、63、94A、129、163番目のAsn残基を含む配列がsequonとなっていた。これらのsequonもすべて、H3N2ウイルス同様、プロトタイプ株の中でCand1、Cand2、Cand3のいずれかに由来していた。したがって、HA遺伝子配列上のCand1、Cand2、Cand3の存在はインフルエンザウイルスがヒトの間で持続的に流行するために重要である可能性を示唆している。

図 7 ヒトH1N1インフルエンザウイルスHAの糖鎖付加部位のコドンの変遷
データは各年代で多数をしめた結果である。1977年に再出現する前のH1N1ウイルスの情報は少ないので、図はモザイク状になっている。
我々は、様々な亜型のインフルエンザウイルスのHA遺伝子を大規模に解析し、糖鎖を獲得するための潜在的ポテンシャルを比較した。sequonの数はHA亜型によって違いはなかったが、Cand1およびCand2の数は亜型によって有意に異なることがわかった。したがって、自然宿主で保持されているインフルエンザウイルスが、新型インフルエンザウイルスとしてヒトの間で流行を始めた場合、糖鎖付加を伴う抗原変異をする能力はHA亜型によって異なることが示唆された。
今回紹介した手法はさらに、それぞれの塩基の置換確率の違いや獲得したsequonに実際に糖鎖付加が起こるための条件等、様々な細胞生物学的現象を考慮する必要性がある。しかしながら、我々のアプローチはヒトに新型インフルエンザウイルスによるパンデミックが起こった場合、そのウイルスが持続的に流行するかどうかを予測する方法の一助となることが期待される17。