Sep. 15, 2000
インフルエンザウイルスとそのグリコレセプター:ヒトおよび動物インフルエンザウイルスの宿主域変異(2000 Vol.04, A7)
鈴木 康夫
氏名:鈴木 康夫
静岡県立大学・薬学部・生化学教室
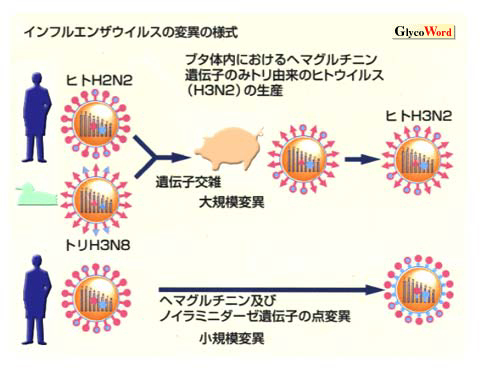
A型インフルエンザウイルスは、ヒト、ブタ、ウマ、海洋哺乳動物、トリなど様々な動物種から分離される。野生の水トリからは全ての異なる亜型(H1〜 H15、 N1〜 N9)が分離されている。我々は、ヒト、ブタ、ウマ、水トリインフルエンザA型ウイルスのへマグルチニンは、いずれも糖タンパク質および糖脂質に含まれるシアリルラクト系 IおよびII型糖鎖 (SAα2-3(6)Galβ1-3(4)GlcNAcβ1-) を共通のレセプター分子として認識していることを明らかにした。
我々はさらに、トリおよびウマから分離されるウイルスは末端のSialic acid α2-3Gal(SA2-3Gal)結合を、ヒトから分離されるウイルスはSA2-6Gal結合を強く認識していることを見出した。インフルエンザウイルスは末端のシアル酸分子種(Neu5Ac, Neu5Gc, 9-O-Ac-Neu5Ac)も識別している。
我々は、初めて、様々な動物種におけるシアル酸の分布はインフルエンザウイルスの宿主域に影響を与える事実を見出した。ブタ気道上皮細胞はトリ(SA2-3Gal)とヒト(SA2-6Gal)のインフルエンザウイルスに対するレセプターを持っている。ウマの場合、Neu5Ac2-3Galではなく、Neu5Ac2-6Galに対して結合性を示すウイルスはウマに感染出来ない。しかし、Neu5Gc2-3Galに結合できるウイルスはウマに感染可能である。ウマの気道上皮細胞には、Neu5Gc2-3Galが多く存在するが、この特性はウマインフルエンザウイルスの増殖に極めて重要である。Neu5Gc2-3Gal は、カモの小腸におけるトリインフルエンザウイルスの増殖に極めて重要である。Neu5Gc2-3Galは、カモの腸管のクリプト細胞に主に存在することを特異抗体により明らかにした。これらの結果は、クリプト細胞のNeu5Gcはカモの腸管におけるインフルエンザウイルスの増殖能と深い関連があることを示していた。
ヒトインフルエンザウイルスはMDCK細胞で増殖させたSA2-6Galに対して高いレセプター認識特異性を示すが発育鶏卵しょう尿膜培養した場合、ウイルス株により違いがあるが、SA2-3Galへの認識特異性がシフトすることを見出した。この変異は、へマグルチニン分子内のただ一つのアミノ酸置換、226Leu →Glnにより生じた。この結果から、へマグルチニン分子内のアミノ酸226はレセプター糖鎖内シアル酸結合様式(2-3, 2-6)の識別に関わるものであることが解った。発育鶏卵しょう尿膜にはSA2-6Galが欠如しており、SA2-3Galが主に存在する。
これらの結果は、インフルエンザウイルスは、宿主の受容体シアロ糖鎖のシアル酸結合様式(2-3、2-6)によって進化上の選択を受け、その結果、宿主の壁を越える可能性があることを示していた。
我々は、Neu5GcとNeu5Acとの識別には、へマグルチニン分子内の、143,155,158、さらに228番目のアミノ酸が重要であることも明らかにしている
References
- Toshihiro Ito, Yasuo Suzuki, Ayato Takada, Ayumi Kawamoto, Koichi Otsuki, Hiroyuki Masuda, Takashi Suzuki, Hiroshi Kida, Yoshihiro Kawaoka : Differences of sialic acid-galactose linkages in the chicken egg amnion and allantois influence human influenza virus receptor specificity and variant selection. J. Virol. 71(4), 3357-3362, 1997
- Takashi Suzuki, Goh Horiike, Yasuhiro Yamazaki, Kaoru Kawabe, Hiroyuki Masuda, Daisei Miyamoto, Masao Matsuda, Shin-Ichiro Nishimura, Tatsuya Yamagata, Toshihiro Ito, Hiroshi Kida, Yoshihiro Kawaoka Yasuo Suzuki : A Swine Influenza virus strains recognize sialylsugar chains containing the molecular species of sialic acid predominantly present in the swine tracheal epitherium. FEBS Lett. 404, 192-196, 1997
- Katsuhiko Sato, Goro Hanagata, Makoto Kiso, Akira Hasegawa, Yasuo Suzuki : Specificity of N1 and N2 sialidase subtypes of human influenza A virus for natural and synthetic gangliosides. Glycobiology, 8 (6), 527 - 532, 1998
- Hiroyuki Masuda, Takashi Suzuki, Yoshiyuki Sugiyama, Goh Horiike, Kohji Murakami, Daisei Miyamoto, Kazuya I-P. Jwa Hidari, Toshihiro Ito, Hiroshi Kida, Makoto Kiso, Kyoko Fukunaga, Masanobu Ohuchi, Tetsuya Toyoda, Akira Ishihama, Yoshihiro Kawaoka, Yasuo Suzuki : Substitution of amino acid residue in influenza A virus hemagglutinin affects recognition of sialyl-oligosaccharides containing N-glycolylneuraminic acid. FEBS LETT. 464, 71-74, 1999
- Kuniho Nakata, Chao-Tan Guo, Motoko Matsufuji, Akihiro Yoshimoto, Masanori Inagaki, Ryuichi Higuchi, Yasuo Suzuki : Influenza A virus- binding activity of glyceroglycolipids of aquatic bacteria. J. Biochem. 127, 191-198, 2000
- Yasuo Suzuki, T. Ito, O. Gao, K. Masuda, C-T. Guo, K. Hidari, D. Miyamoto, T. Suzuki, Y. Kawaoka : Host madiated variation of influenza viruses. Glycoconjugate J.16 (4/5), S39, 1999
- Darwin Kobasa, Shantha Kodihalli, Ming Luo, Maria R. Castrucci,Isabella Donateli, Yasuo Suzki, Takashi Suzuki, Yoshihiro Kawaoka : Amino acid residues contributiong to the substrate specificity of theinfluenza A virus neuraminidase. J. Virol. 73, 6743-6751, 1999
- Yasuo Suzuki, Toshihiro Ito, Takashi Suzuki, Robert E. Holland, Thomas M. Chambers, Makoto Kiso, Hideharu Ishida, Yoshihiro Kawaoka : Sialic acid species as a determinant of the host range of influenza A viruses. J. Virol. in press, 2000
- Toshihiro Ito,Yasuo Suzuki, Takashi Suzuki, Ayato Tanaka, Taisuke Horimoto, Krisna Wells, Hiroshi Kida, Koichi Otsuki, Makoto Kiso, Hideharu Ishida, Yoshihiro Kawaoka: Recognition of N-glycolylneuraminic acid linked to galactose by alpha 2-3 linkage is associated with the intestinal replication ofinfluenza A virus in ducks. J. Virol., 74 (19) 9300-9305, 2000
