
氏名:赤瀬 幸子
創価大学 工学部 生命情報工学科を卒業し、現在、創価大学大学院 工学研究科 博士前期課程で、木下聖子教授のもと糖鎖情報科学について学んでいます。糖鎖遺伝子から糖鎖合成を予測できるWebツール「GlycoSim」の開発に従事し、FlyGlycoDBに登録されている遺伝子を用いたショウジョウバエのN型糖鎖合成シミュレーションを行なっています。

氏名:安形 清彦
筑波大学大学院 生物科学研究科で学位取得後、La Jolla Cancer Research Foundation(現Sanford Burnham Prebys Medical Discovery Institute)にて、福田穰教授のもとで糖鎖生物学の研究について学ぶ。現所属(産総研)では、糖鎖遺伝子の発現解析、糖鎖の新しい機能の解析や糖鎖の応用について研究するとともに糖鎖DB(ACGG-DB)の開発に参画しています。
本シリーズの第3回では、糖鎖関連遺伝子のデータベースについて説明いたします。糖鎖の合成に関わる遺伝子(糖鎖関連遺伝子, glycogene)には、糖ヌクレオチド合成酵素・糖ヌクレオチドトランスポーター・糖転移酵素などの合成のための関連分子や分解のために必要なグリコシダーゼの遺伝子が含まれています。ヒトでは約300遺伝子ほどが糖鎖の合成や分解に関与していますが、生物の進化において数や構成している遺伝子の多様性も異なっています。ここでは、先ず糖鎖関連遺伝子の研究に有用と思われるデータベース(DB)について紹介し、筆者らが公開しているデータベース(GGDBとFlyGlycoDB)の特徴についても紹介します。
糖鎖研究はこれまでの糖鎖の構造や生合成から、微生物学、発生生物学、免疫学や腫瘍学など多分野から糖鎖の機能を明らかにする研究が増えています。そこで、これまで糖鎖研究を実施していなかった研究者にとって、様々な情報を集約したDBは糖鎖研究を進める上で論文を読むのと同様に役に立つと考えられます。仮に糖鎖遺伝子名が論文や解析結果に出て来た場合、糖鎖遺伝子についての手掛かりとして一般的な遺伝子やタンパク質のDBや知識ベース(knowledge base, KB)も利用できますが、糖鎖遺伝子の情報収集のための手段としてここで紹介する糖鎖関連DBも公開されており、それぞれのDBの特徴を活かした利用が有効です。
例えば、糖鎖遺伝子のcDNAやゲノム構造などの配列の取得には、gene、Ensembl、CAZyがあります。特にCAZyは様々な生物種の糖鎖遺伝子を網羅しており、生物種間の比較や進化的な解析に有用です。病気との関係を調べるには、OMIM(https://www.ncbi.nlm.nih.gov/omim, https://omim.org)やHuman Protein Atlasが有用です。Human Protein Atlasでは癌組織での糖鎖遺伝子の発現量を見ることができます。タンパク質としての解析には、KEGG、UniProtKB、GlyGen、GeneCardsなどが用いられ、パスウエイ、質量分析やインタラクトームの解析結果にアクセスすることができます。
ヒトではこれまでに約300個の糖鎖関連遺伝子がクローニングされています。これらのうち糖鎖合成に関わる遺伝子に着目したデータベースがGlycoGene Database(GGDB)で、日本糖鎖科学総合データベース(JCGGDB)の一つとして公開されました。現在では表示方法などインターフェースや記載内容などにも変更を加えた最新版をACGG内で公開していますが(https://acgg.asia/db/ggdb/, Narimatsu et al. 2017)、ここではその特徴について紹介いたします。
※ GGDBはACGGからだけではなく、日本糖鎖科学コンソーシアム(JCGG)やGlyCosmosポータルからも訪れることが出来ます。現在、221個の糖鎖合成に関与する糖鎖遺伝子をリストしていますが、トップページでは糖鎖遺伝子がアルファベット順に並べられ酵素反応やキーワードが簡単に見ることができます(図 1)。
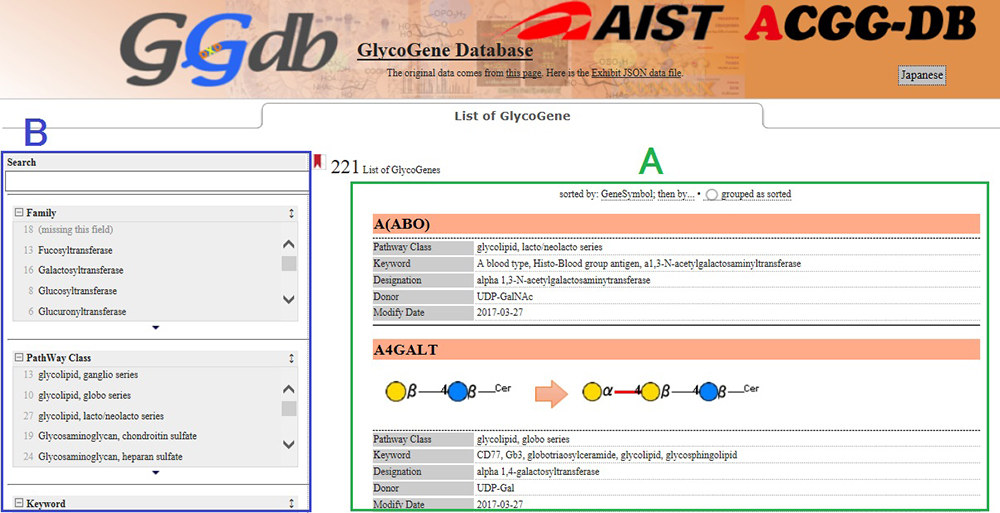
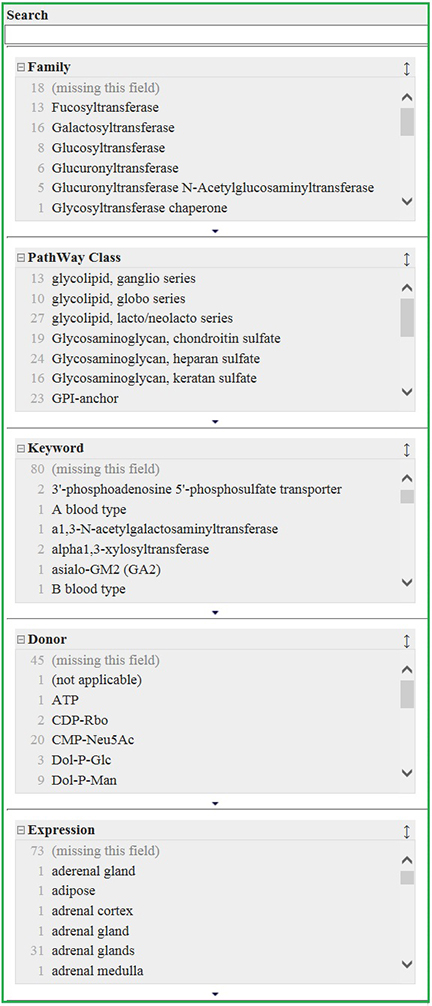
また、検索モードとして、テキスト検索に加えファセット検索を用意してありますので絞り込み検索が簡単にできます(図 2)。
次に選択した糖鎖遺伝子のページを説明します。
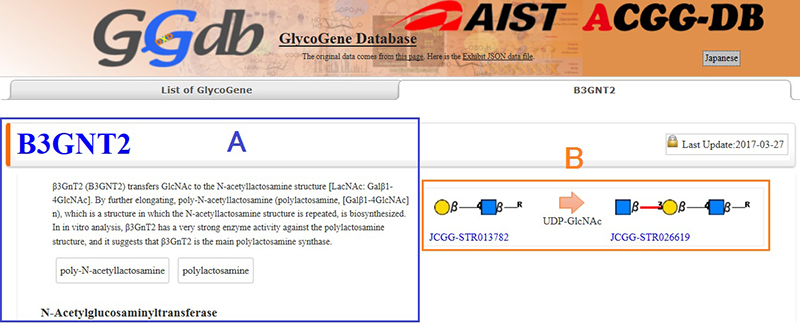
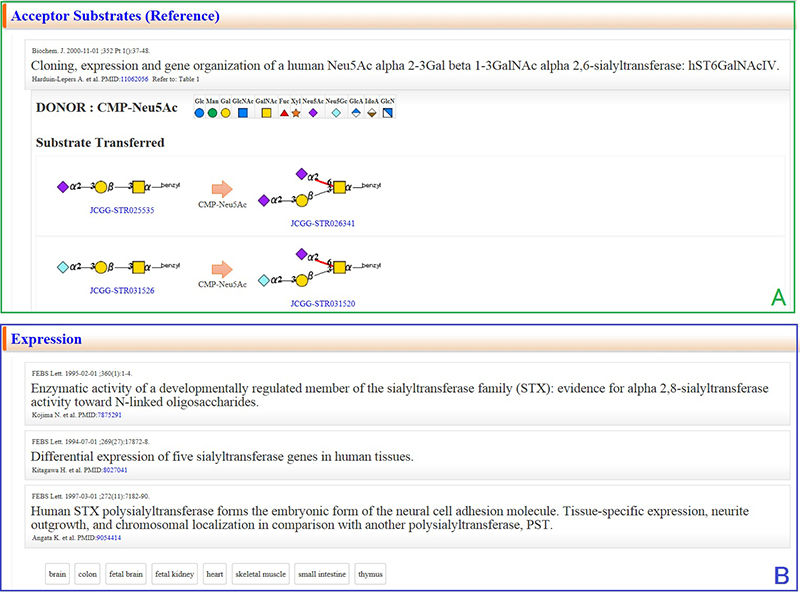
選択された糖鎖遺伝子の各ページでは、サマリーと主な酵素反応が前駆体、基質(糖ヌクレオチド)と反応産物が図で示されています(図 3)。GGDBを含めたACGG-DBの共通の特徴は、全てのデータベースが糖鎖の構造という共通言語(JCGG-STR)を用いているということです。上述の一般的なDBでは文字による記載のみであるのに対して、直感的に反応産物をイメージできることが特徴の一つです。サマリーは産業技術総合研究所(産総研)の糖鎖研究者がキュレーションを担当しており、酵素反応や機能を簡単にまとめてあります。
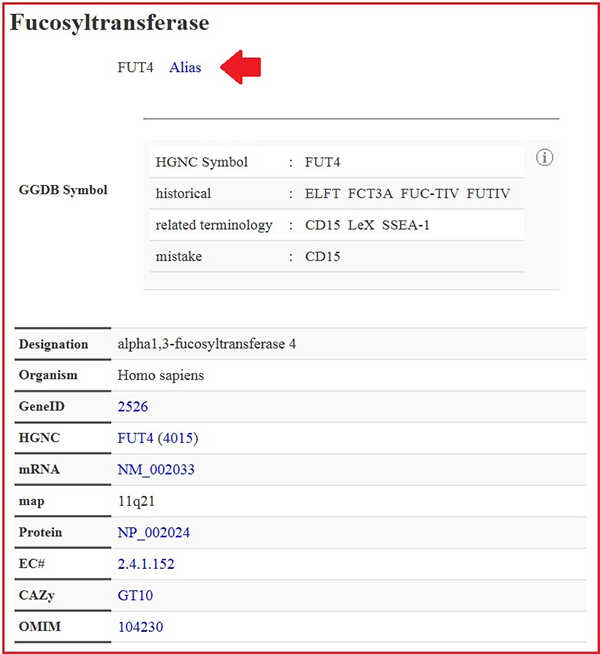
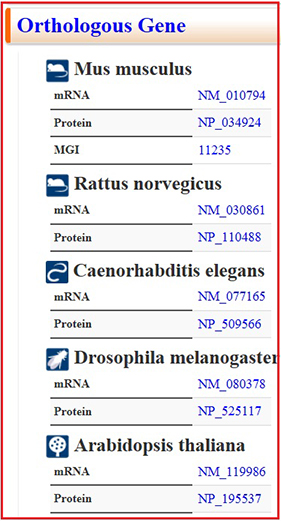
遺伝子名はHGNCでの命名を採用しています。これまでに使用されている通称はエリアス内に収納されています。糖鎖研究の長い歴史の中で糖鎖遺伝子には様々な名前がつけられています。例えばFUT4遺伝子は生成物であるCD15に因んでCD15遺伝子と呼ばれていたので、エリアス内に別名として入れてありますが、混乱を避けるために今後の使用は避けるべきで、参考のために記載しています(図 4)。
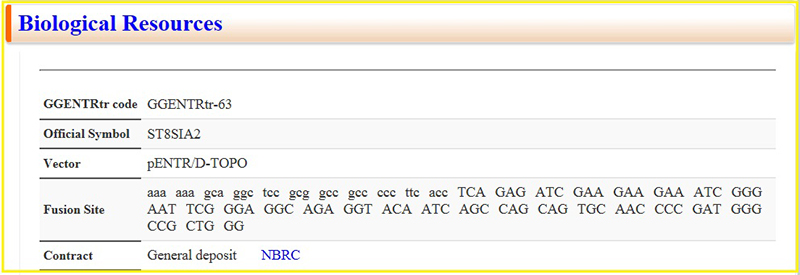
中段以降では、相同遺伝子(図 5)、酵素反応やmRNA発現(図 6)、リーソース(図 7)などの情報を取得することができます。
2019年現在でも、新しい糖鎖遺伝子あるいは糖鎖遺伝子の活性や機能が報告されていますので、サマリーの更新や酵素反応の改定も定期的に必要です。また、糖鎖合成パスウエイに関わる糖鎖遺伝子の変異や発現量の情報は、糖鎖の異常を伴う疾病や癌などの研究にも有用と考えられます。今後はGlyCosmos内での連携を進めることによって、他のDBとの横断的な利用を開発する予定です。
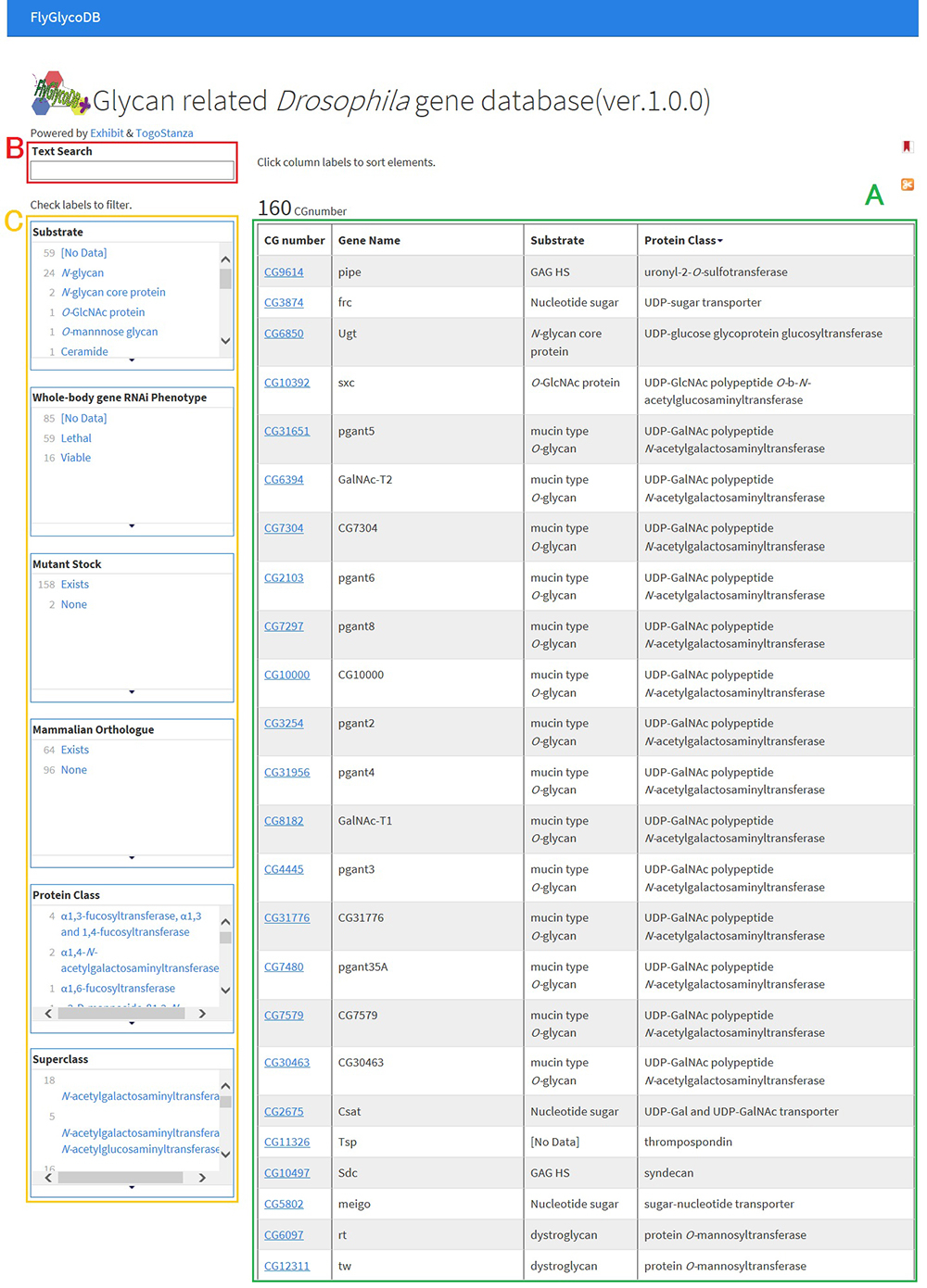
キイロショウジョウバエ(学名: Drosophila melanogaster、以下ショウジョウバエ)の糖鎖関連遺伝子のデータベースであるFlyGlycoDBは、http://fly.glycoinfo.org/から利用することができます。ショウジョウバエは、糖鎖研究に限らず、多くの生物学的研究において、古くからモデル生物として用いられてきました。ショウジョウバエの遺伝学的実験結果をまとめたデータベースFlyBase(Thurmond et al. 2019)は、ショウジョウバエで発見されている全遺伝子を格納していますが、その中から糖鎖関連遺伝子を抽出するのは難しく、ショウジョウバエの糖鎖関連遺伝子のみに着目したデータベースは存在していませんでした。そこで、糖鎖研究の視点から利用しやすいデータベースとして、FlyGlycoDBが開発されました。FlyGlycoDBでは現在、RNAiサイレンシングを用いた解析によって活性が確認された遺伝子(Nishihara et al. 2007, 2010; Yamamoto et al. 2015)、および糖関連酵素データベースCAZy(Lombard et al. 2014)から取得した遺伝子など、計160の機能同定されたショウジョウバエ糖関連遺伝子が登録されています。FlyGlycoDBのトップページを図 8に、各遺伝子のエントリーページを図 9に示します。
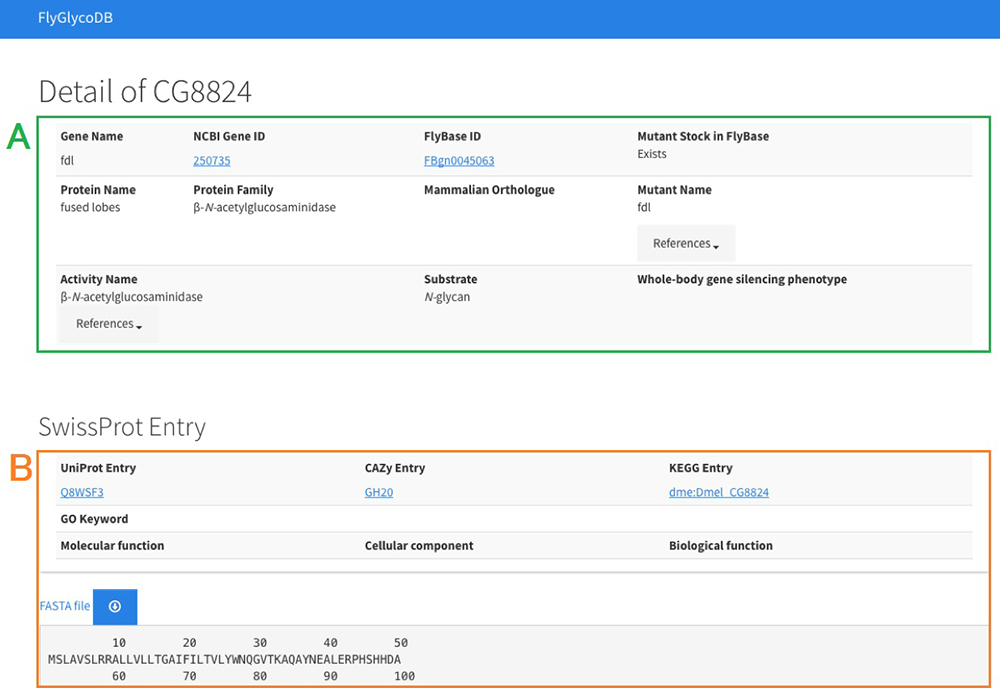
FlyGlycoDBはセマンティックウェブ化されているため、外部のデータベースとの連携を容易に行うことができます。そのためエントリーページでは、選択された遺伝子について、FlyGlycoDBに保存されている情報だけでなく、SwissProtの情報も表示できるようになりました。
また、2019年8月1日より、アップデートされたFlyGlycoDBが、GlyCosmosから利用できます。GlyCosmos Data Resources下にて、分類された全166エントリーのショウジョウバエ糖関連遺伝子が閲覧可能です。