 |
小胞体は、全タンパク質のおよそ1/3のタンパク質が生合成される場であり、膜タンパク質や分泌タンパク質が新規合成される。小胞体におけるタンパク質の翻訳後修飾として、PDI (protein disulfide isomerase)ファミリーが媒介するジスルフィド結合形成や、Asn-X-Ser/Thr (X≠Pro)からなるコンセンサス配列のAsnに結合するN型糖鎖修飾がある。N型糖鎖は、小胞体を通過するおよそ80 %のタンパク質を修飾する。これは、3つのグルコース、9つのマンノース、2つのN-アセチルグルコサミンからなり(G3M9と呼ぶ)、図、左からA、B、C鎖と呼ぶ。N型糖鎖は、小胞体におけるタンパク質の構造形成と分解に密接な関連がある(QS-A00参照) (1)。
高等動物においては、タンパク質に付加されたN型糖鎖から、2つグルコースがトリミングされたGM9型糖鎖をレクチンシャペロンであるCalnexin(CNX)やCalreticulin(CRT)が認識し、タンパク質の構造形成を促す。さらにグルコースがトリミングされM9型になった際にタンパク質部分が構造形成していると、タンパク質は分泌経路へと進む。構造形成していない場合は、UGGTに認識され、M9 型N型糖鎖にグルコースが1つ再付加されることによってGM9型糖鎖となり、CNXやCRTの基質となり、タンパク質の構造形成が再び促される。このグルコースの切除と再付加に依存した構造形成サイクルをCNX/CRTサイクルと言う(QS-A01参照)。ただし、このシステムは、酵母ではあまり機能的でない。
しかし、なかなか構造形成できない構造形成不良タンパク質は、小胞体の負荷になるため、分解除去する必要がある。糖鎖依存的な分解経路では、糖タンパク質のM9型糖鎖のマンノースが切除され、M8B型糖鎖へとトリミングされた後に、M8B型糖鎖からM7、M6、M5型糖鎖へとトリミングされる。最外部のC鎖のマンノースが切除されると、レクチン分解因子である酵母Yos9、高等動物OS9/XTP3Bに構造形成不良タンパク質は認識されるようになり、それは小胞体から細胞質へ逆行輸送され、N型糖鎖が外され、ユビキチンープロテアソームシステムによる分解へと導かれる(QS-A03、A04参照) (1)。
酵母において、小胞体におけるマンノースのトリミングはM9からM8BはMns1が無作為的に行い、M8Bからはジスルフィド結合を含んだ複合体であるHtm1-S-S-Pdi1複合体が遂行する。つまり律速段階は1つであり、Htm1複合体が分解に導くタンパク質を決定する。一方で高等動物において、小胞体でのマンノーストリミングを行う候補分子は、MAN1B1、EDEM1、EDEM2、EDEM3の4つが過去には挙げられていた。これらの分子は、すべてMannosidase homology domainを持つ。しかし、内在性MAN1B1は、ゴルジ体に局在することが示された(2)。そして、EDEM1、EDEM2、EDEM3の細胞内における内在性遺伝子の機能解析やin vitro酵素活性が調べられ、M9からM8BへのトリミングはEDEM2が遂行し(3,4)、M8BからのトリミングはEDEM3とEDEM1が遂行する(主にはEDEM3)ことが明らかにされた(4,5)。ただし2005年の報告において、EDEM2は単独では強いin vitro酵素活性を発揮することができなかったため、その後EDEM2のパートナー分子としてTXNDC11が同定されている。TXNDC11は5つのチオレドキン様ドメインを持ち、C末端には、Coiled-coilドメインを有す。EDEM2のC558とTXNDC11のC692がジスルフィド結合を形成し、EDEM2-S-S-TXNDC11の複合体として酵素活性を発揮する(3)。
分解基質は、おそらくタンパク質の疎水性面が外側にきて、タンパク質のゆらぎが大きいものから選別されていくと考えられる。この選別過程において、酵母では、M8Bからのマンノースのトリミングによって分解基質を1段階で選別するが、高等動物ではM9→M8B、M8Bからのマンノーストリミングの2段階から成っているため、高等動物は酵母に比べてより慎重にどのタンパク質を分解するかということを決定していると考えられる。
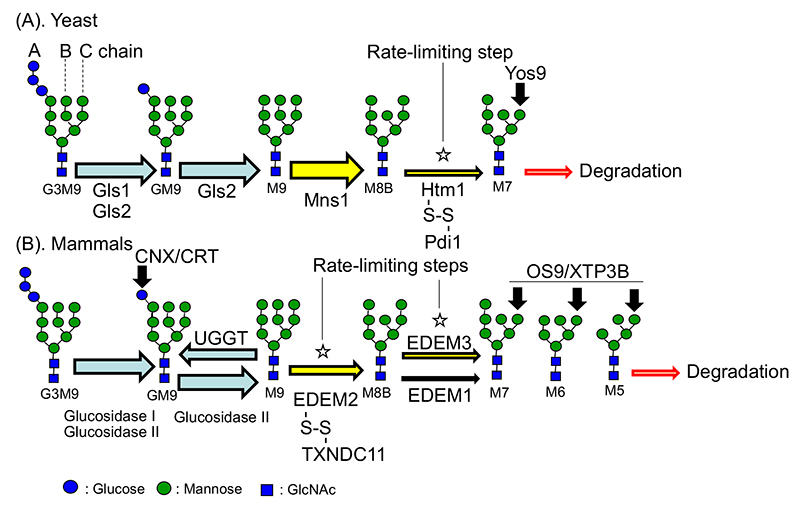
図
(A) N型糖鎖の鎖は、左からA、B、C鎖と呼ぶ。酵母において、G3M9→G2M9はGls1が、G2M9→GM9→M9はGls2がグルコースのトリミングを行う。M9からはMns1が無作為にマンノースのトリミングを遂行する。M8Bからのトリミングは、Htm1-S-S-Pdi1複合体が遂行する。C鎖のマンノースがトリミングされると、Yos9に認識されるようになり、分解へと導かれる。(B). 高等動物では、G3M9→G2M9はGlucosidase Iが、G2M9→GM9→M9はGlucosidase IIがトリミングを遂行する。GM9型糖鎖は、CNX、CRTによって認識され、タンパク質部分の構造形成が促進される。タンパク質部分が構造形成すると、分泌経路へと進むが、構造形成できていないと、UGGTによってグルコースが再付加され、CNX、CRTの基質になり、構造形成が再び促進される。構造形成できない構造不良タンパク質は、M9→M8BへとEDEM2-S-S-TXNDC11複合体によってトリミングされた後、M8B→M7、M6、M5へとEDEM3、EDEM1によってトリミングされる。C鎖のマンノースがトリミングされると、OS9、XTP3Bに認識されるようになり、分解へと導かれる。高等動物の場合、分解基質を選別する段階が、M9→M8B、M8Bからのマンノーストリミングの2段階から成っているため、1段階からなる酵母に比べてより慎重に分解すべき基質を選別しているといえる。
蜷川 暁(神戸大学バイオシグナル総合研究センター)
| References |
| (1) |
Ninagawa S, George G, Mori K: Mechanisms of productive folding and endoplasmic reticulum-associated degradation of glycoproteins and non-glycoproteins. Biochim. Biophys. Acta Gen. Subj. 1865, 129812, 2021 |
| (2) |
Pan S, Wang S, Utama B, Huang L, Blok N, Estes MK, Moremen KW, Sifers RN: Golgi localization of ERManI defines spatial separation of the mammalian glycoprotein quality control system. Mol. Biol. Cell 22, 2810-2822, 2011 |
| (3) |
George G, Ninagawa S, Yagi H, Saito T, Ishikawa T, Sakuma T, Yamamoto T, Imami K, Ishihama Y, Kato K, Okada T, Mori K: EDEM2 stably disulfide-bonded to TXNDC11 catalyzes the first mannose trimming step in mammalian glycoprotein ERAD. eLife 9, e53455, 2020 |
| (4) |
Ninagawa S, Okada T, Sumitomo Y, Kamiya Y, Kato K, Horimoto S, Ishikawa T, Takeda S, Sakuma T, Yamamoto T, Mori K: EDEM2 initiates mammalian glycoprotein ERAD by catalyzing the first mannose trimming step. J. Cell Biol. 206, 347-356, 2014 |
| (5) |
George G, Ninagawa S, Yagi H, Furukawa JI, Hashii N, Ishii-Watabe A, Deng Y, Matsushita K, Ishikawa T, Mamahit YP, Maki Y, Kajihara Y, Kato K, Okada T, Mori K: Purified EDEM3 or EDEM1 alone produces determinant oligosaccharide structures from M8B in mammalian glycoprotein ERAD. eLife 10, e70357, 2021 |
2023年 6月15日
|
|---|






