この作品はクリエイティブ・コモンズ 表示 4.0 |
 |
|---|
糖転移酵素の基質タンパク質選択性 | ||||||||||||||||||||||||||||||||
 |
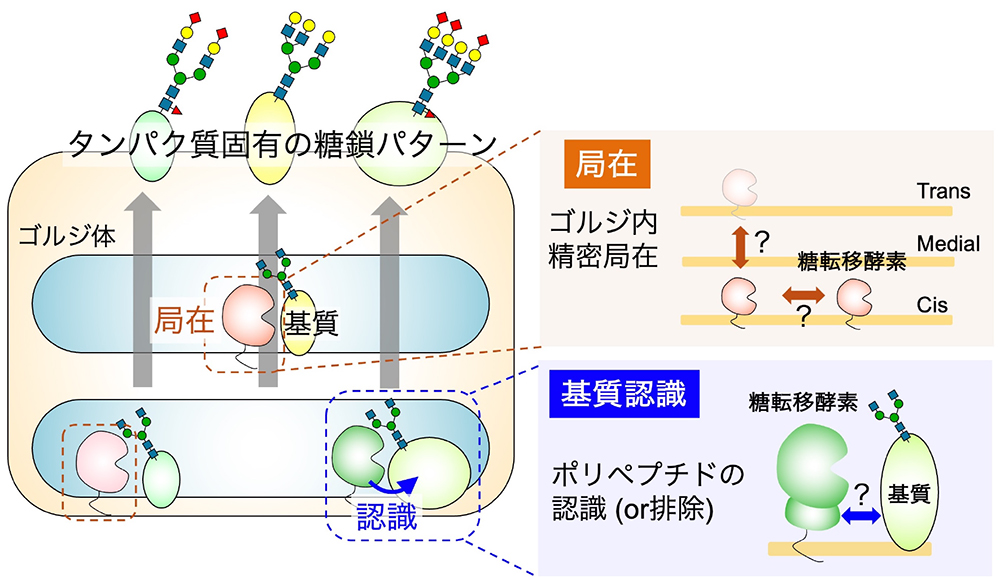
動物の糖鎖の多くはタンパク質に付加された形で存在している。それらの分類としては、糖鎖構造や付加するアミノ酸によって、N型糖鎖、O型糖鎖(コア構造によりさらにO-GalNAc、O-Man、O-Fuc、O-GlcNAc、O-Galなどに分類)、グリコサミノグリカン(GAG)、Glycosylphosphatidylinositol (GPI)の4つのクラスが存在する。そして周知の事実として、付加されている糖鎖の構造はタンパク質ごとに異なる。クラスの違う糖鎖が異なるタンパク質に付加されることについては、仕組みが明確になっているケースが多く、ほとんどの場合、各糖鎖の生合成の一番初めのステップ、すなわちアミノ酸への糖(鎖)の転移、を触媒する酵素の基質認識により決まる。例えばO-Fuc型が付加されるかどうかは、protein O-fucosyltransferase (POFUT)1,2が基質タンパク質のEpidermal growth factor (EGF)ドメインやthrombospondin type 1 repeat (TSR)ドメインを認識することによるし(1)、GPIが付加されるかどうかは、GPI transamidase complexがC末端周辺のGPI付加配列を認識して決める(2)。このように、各糖鎖クラスの付加の有無自体については、酵素の認識に必要なアミノ酸配列やドメインに関する研究が進んでいる。 また、根元の糖(鎖)が付加された後の、糖鎖が伸長していく部分については、糖転移酵素によって基質となる糖鎖が認識されてその先に糖が伸ばされていくが、各酵素によって非還元末端側の糖鎖構造に対する特異性は異なることが知られている。例えば、GlcAを転移するGlcAT-P、GlcAT-Sに関しては、3本鎖のN型糖鎖の還元末端に結合する3つのNアセチルラクトサミン(Galβ1-4GlcNAc)構造に対して、それぞれ異なる反応性を示すことが知られている(3)。 一方、タンパク質ごとに糖鎖の発現プロファイルが異なること自体はわかっているが、それを決める法則性についてはよくわかっていない。例えば、イムノグロブリンG(IgG)は2分岐N型糖鎖しか発現していないが、エリスロポエチンなどでは4分岐が多いことが知られている。またこのようなタンパク質間の糖鎖構造プロファイルの違いは、それらのタンパク質が同じ細胞由来であっても生じる。さらには、O-GalNAc型の伸長・分岐・シアリル化、GAGの長さの違いや硫酸の程度やポジション、などにも同様のことが言える。特に、糖鎖の末端構造(例えばシアル酸、ルイス型抗原、硫酸化など)の存在の度合いがタンパク質ごとに異なる仕組みについては、不明な点が多い。 対象の糖鎖が付加されるタンパク質が極めて限定的な場合は、そのタンパク質が持つ特定のドメインやモチーフを糖転移酵素が認識することでそれを可能にしている。例えば、N型糖鎖末端のポリシアル酸はほとんどがneural cell adhesion molecule (NCAM)に付加されているが、それはポリシアル酸の生合成酵素ST8SIA2および ST8SIA4がNCAMのFibronectin type III様ドメインの酸性領域を特異的に認識することによる(4)。また最近では、FUT9が作るLewis X修飾がLAMP1上の特定のアミノ酸配列を認識して付加することなどもわかってきた(5)。他にも、生合成の欠損が筋ジストロフィーの原因になることでよく知られるCoreM3型O-Man糖鎖は、α-dystroglycanの特異的なアミノ酸残基に限定的に発現しているが(6)、その背景としてPOMGNT2が糖鎖近傍のアミノ酸配列を認識する仕組みが報告されている(7,8)。 一方で、タンパク質の選択性があること自体はグライコーム解析でわかっているものの、多くのタンパク質に存在する構造、例えばN型糖鎖の分岐、N型やO型の末端シアル酸などの修飾、GAGの硫酸化、などについては、現在のところ、そのタンパク質ごとの違いを生む明確な規定因子が不明な場合がほとんどである。タンパク質上の糖鎖構造の成熟は、小胞体やゴルジ体を糖タンパク質が通過する過程で、当該タンパク質が様々な酵素と順序よく出会うことで成立する。それを考えれば、特定の糖鎖が当該タンパク質上に付加されるかどうかを決める要因は、大別すると、作られる酵素と基質が出会うかどうか(局在の問題)と、出会った場合に実際の糖転移反応が起こるかどうか(認識の問題)、の2つが存在すると考えられる。実際には、ここにさらに酵素の発現量、ドナー基質となる糖ヌクレオチドの濃度、基質タンパク質のフォールディング状態、なども重要になってくるが、本章では上記の局在と認識の2点に絞って考えたい。 糖転移酵素の局在の詳細については、別の章を参照していただきたい(”糖転移酵素の基質タンパク質選択性”の頁参照)が、動物細胞では、糖転移酵素はゴルジ体のcis, medial, transの槽の中のさらに細かいゾーンに分かれて偏在していると考えられる。当然のことながら、酵素と基質が出会わなければ糖転移反応は起きないので、酵素の局在は糖鎖の合成に極めて重要なファクターであるが(9)、その局在を規定する仕組みについては不明な点が多い。 一方、酵素による基質糖タンパク質の認識についても、従来の生化学的解析は遊離糖鎖を基質とした酵素活性測定系を中心としてきた背景があり(10,11)、現時点においても情報がまだ少ない。当然、基質タンパク質と酵素の立体障害は糖転移の有無を決める大きな要因の一つであり、AlphaFoldを始めとする精度の高い構造予測が利用できる現在にあっては(12)、個々の糖タンパク質と糖転移酵素群との立体障害についての解析も可能になってくるであろう。一方、糖転移酵素の中には、タンパク質への糖の転移に不可欠な機能ドメインを触媒ドメインとは別に持っているものも存在することがわかってきた(13,14)。今後、触媒ドメイン以外の機能ドメインによる基質タンパク質の選別機構があるかどうかが新たなトピックの一つになる可能性がある。 一方で、こうした複合体形成については、細胞株を用いた過剰発現の系で証拠が得られている場合が多いため、内在性の酵素が組織中でどのように複合体を形成しているのかはいまだ十分な証拠が得られていないケースもある。近年、CRISPRを用いたゲノム編集、またTurboIDなどを始めとする近接標識などの技術が進展しており、内在性酵素へのタグのノックインや、IP(免疫沈降)-ウエスタンブロッティングに依らない相互作用の検出などが可能になってきている。これらの技術の進展とともに、様々な細胞において内在性の酵素がどのような複合体状態で存在しているのかを明らかにすることが、糖鎖生合成の全貌を理解する上で必要な次のステップになると考えられる。
木塚 康彦(岐阜大学 糖鎖生命コア研究所)、
|
|||||||||||||||||||||||||||||||
| References | |
|---|---|
| (1) | Holdener BC, Haltiwanger RS: Protein O-fucosylation: structure and function. Curr. Opin. Struct. Biol. 56, 78-86, 2019 |
| (2) | Kinoshita T: Biosynthesis and deficiencies of glycosylphosphatidylinositol. Proc. Jpn. Acad. Ser. B Phys. Biol. Sci. 90, 130-143, 2014 |
| (3) | Yagi H, Yamada K, Ohno E, Utsumi M, Yamaguchi Y, Kurimoto E, Takahashi N, OKa S, Kawasaki T, Kato K: Development and Application of High Performance Liquid Chromatography Map of Glucuronyl N-glycans. Open Glycoscience 1, 8-18, 2008 |
| (4) | Sato C, Kitajima K: Polysialylation and disease. Mol. Aspects Med. 79, 100892, 2021 |
| (5) | Saito T, Yagi H, Kuo CW, Khoo KH, Kato K: An embeddable molecular code for Lewis X modification through interaction with fucosyltransferase 9. Commun. Biol. 5, 676, 2022 |
| (6) | Yagi H, Kuo CW, Obayashi T, Ninagawa S, Khoo KH, Kato K: Direct Mapping of Additional Modifications on Phosphorylated O-glycans of alpha-Dystroglycan by Mass Spectrometry Analysis in Conjunction with Knocking Out of Causative Genes for Dystroglycanopathy. Mol. Cell. Proteomics 15, 3424-3434, 2016 |
| (7) | Imae R, Kuwabara N, Manya H, Tanaka T, Tsuyuguchi M, Mizuno M, Endo T, Kato R: The structure of POMGNT2 provides new insights into the mechanism to determine the functional O-mannosylation site on alpha-dystroglycan. Genes Cells 26, 485-494, 2021 |
| (8) | Yang JY, Halmo SM, Praissman J, Chapla D, Singh D, Wells L, Moremen KW, Lanzilotta WN: Crystal structures of beta-1,4-N-acetylglucosaminyltransferase 2: structural basis for inherited muscular dystrophies. Acta Crystallogr. D Struct. Biol. 77, 486-495, 2021 |
| (9) | Welch LG, Munro S: A tale of short tails, through thick and thin: investigating the sorting mechanisms of Golgi enzymes. FEBS Lett. 593, 2452-2465, 2019 |
| (10) | Brockhausen I, Carver JP, Schachter H: Control of glycoprotein synthesis. The use of oligosaccharide substrates and HPLC to study the sequential pathway for N-acetylglucosaminyltransferases I, II, III, IV, V, and VI in the biosynthesis of highly branched N-glycans by hen oviduct membranes. Biochem. Cell Biol. 66, 1134-1151, 1988 |
| (11) | Takamatsu S, Korekane H, Ohtsubo K, Oguri S, Park JY, Matsumoto A, Taniguchi N: N-acetylglucosaminyltransferase (GnT) assays using fluorescent oligosaccharide acceptor substrates: GnT-III, IV, V, and IX (GnT-Vb). Methods Mol. Biol. 1022, 283-298, 2013 |
| (12) | Mirdita M, Schütze K, Moriwaki Y, Heo L, Ovchinnikov S, Steinegger M: ColabFold: making protein folding accessible to all. Nat. Methods 19, 679-682, 2022 |
| (13) | Osuka RF, Hirata T, Nagae M, Nakano M, Shibata H, Okamoto R, Kizuka Y: N-acetylglucosaminyltransferase-V requires a specific noncatalytic luminal domain for its activity toward glycoprotein substrates. J. Biol. Chem. 298, 101666, 2022 |
| (14) | Osada N, Nagae M, Nakano M, Hirata T, Kizuka Y: Examination of differential glycoprotein preferences of N-acetylglucosaminyltransferase-IV isozymes a and b. J. Biol. Chem. 298, 102400, 2022 |
2024年 4月17日